식품 빅데이터, 그 의미와 가치
우리 생활 모든 정보가 빅데이터이다
최근 온라인 뉴스 기사에 하루도 빠지지 않고 등장하는 용어가 빅데이터이다. 선뜻 보면 빅데이터가 최근에 떠오른 핫한 용어라 생각할 수 있지만 사실 오래전부터 우리는 이미 빅데이터를 생산하고 있었지만, 그것이 보이지 않아 빅데이터라 부르지 않았을 뿐이다. 생활 빅데이터를 예로 들어보자. 우리는 삼시 세끼 밥을 먹고 잠을 자고 운동도 하고 아프면 병원을 가는 이런 일상들을 반복하면서 라이프로그 정보들을 생산하고 있다. 하지만 생산한다고 해서 데이터가 되는 것이 아니고 그걸 기록하고 축적이 되었을 때 비로소 빅데이터라고 말할 수 있다. 다양한 센서기술들이 탑재된 휴대전화기는 우리 생활 데이터들을 빅데이터 화 시키는 일을 가능케 하고 있으며, 이렇게 축적된 빅데이터를 활용한 산업들이 계속해서 진화하고 있다. 이번 포스팅에서는 수많은 생활 데이터 중에서 먹고 사는 것에 관한 식품 빅데이터에 대해 적어보고자 한다.
식품은 어떤 정보와 가치를 가지고 있나?
식품의 맛 정보
기본적으로 우리가 식품정보라 하면 맛과 영양성분 정보들을 들 수 있다. 맛은 굉장히 주관적인 정보이지만 우리가 맛집을 검색할 때 특정 음식점의 음식 맛을 평가한 블로그 정보들을 보고 찾아가는 경우가 많다. 필자도 맛집 탐방을 취미로 하고 있어서 각종 포털의 블로그 정보들을 활용하고 있다. 이렇다 보니, 많은 음식점에서 블로그 마케팅을 내세워 판매수익을 올리기도 한다. 2013년 외식 트렌드 조사에 따르면, 소비자의 대다수(84.2%)는 모바일기기가 보편화된 후로 외식 생활이 변화했다고 생각하고 있는 것으로 나타났다. 응답자의 53.5%는 모바일 기기를 이용하여 방문할 음식점의 맛 정보들을 수집하여 방문하는 것으로 조사되었다(그림1). 외식문화가 변화하면서 스타트업과 대기업을 막론하고 다양한 기업들이 맛집 앱 시장에 문을 두드리고 있다. 대표적인 애플리케이션으로는 포잉, 다이닝코드, 식신, 망고플레이트들이 있으며 누적 다운로드 10만 이상을 기록하는 성과를 거두고 있다. 이처럼 식품의 맛 정보는 주관적인 정보임에도 불구하고 외식 산업적으로 활용가치가 높은 정보라 할 수 있다.
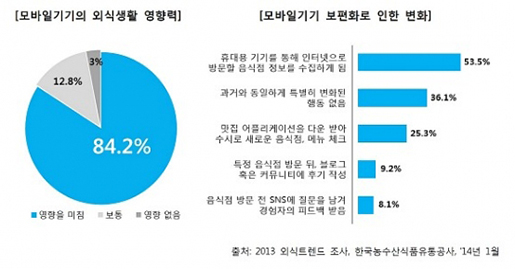
그림1. 식품의 맛 정보, 변화하고 있는 외식 트렌드
(출처 : 한국농수산식품유통공사, 외식 트렌드 조사, 2013)
식품의 영양성분 및 생리활성 정보
식품의 영양성분 정보에 대해 크게 관심이 있는 일반인들은 드물다. 고작 식품에 강조표시되어 있는 sugar free와 low fat 등의 정보만 가지고 본인의 기호에 맞게 구매하는 정도일 것이다. 하지만 식품을 구성하는 영양성분 정보야말로 건강한 삶을 추구하는 인간에게 근본적인 답을 줄 수 있는 정보이고, 구매자는 식품 영양성분 정보에 대해 알 권리가 있다. 모든 식품에 대해 영양성분을 표시할 필요는 없으나 식약처에서는 식품 영양성분 표시에 대한 기준을 제시하고 식품위생법 시행규칙 제6조 제1항에 따라 영양성분을 표시해야 하는 식품의 종류를 정해놓고 있다. 표시 대상 성분은 열량, 탄수화물, 단백질, 지방, 콜레스테롤, 나트륨, 그 밖에 강조표시를 하고자 하는 영양성분으로 크게 7가지를 표시하도록 되어있다.

그림2. 식품 영양성분 예시(출처 : 서울특별시 어린이 식품안전)
국내외적으로 식품의 영양성분 정보는 정부의 식품 데이터베이스에서 제공받을 수 있다. 우리나라의 경우는 식약처에서 구축한 FANTASY DB(http://www.foodsafetykorea.go.kr)에서 확인할 수 있다. 식품별 영양성분 함량과 영양학적 조언 등의 정보들을 포함하고 있으며(그림3), 현재 약 13,713건의 정보가 등록되어 있는 것으로 확인된다.
미국은 USDA DB(https://ndb.nal.usda.gov/ndb)를 만들어 농업과 식품에 대한 정보들을 제공하고 있으며, 유럽의 경우도 EUROFIR DB(http://www.eurofir.org)를 구축하여 유럽 27개국의 식품정보들을 확인할 수 있는 플랫폼을 제공하고 있다. 국가 차원에서 이러한 식품 데이터베이스를 구축하는 이유는 여러 산업과의 연계뿐만 아니라, 신규 사업을 융성하기 위한 취지로 식품정보들을 제공하고 있다. 실제, 미국 기업 중 일부는 USDA DB를 활용하여 헬스케어, 다어어트, 질환 개선을 위한 다양한 애플리케이션을 개발하고 되고 있으며, 대표적으로 HealthWatch 360, CaloryGuard Pro, Nutrition complete 등이 있다.
영양성분 정보가 중요한 이유 중 하나는 대사체 정보를 중심으로 생리활성 정보들과의 연결이 가능하다는 점이다. 예를 들어, 우리나라 전통식품인 김치에 vitamin, carotene, ascorbic acid 등과 같은 성분들이 함유돼 있다고 했을 때, 이러한 정보들을 텍스트마이닝 기법을 활용하여 논문의 생리활성 효능 정보들과 연결하게 되면, 체내에서 식품이 특정 질병에 얼마나 효과적인지를 판단할 수 있는 정보가 될 수 있다(그림4). 이러한 정보들은 건강 기능성 식품 개발에 있어, 건강증진에 도움이 될 수 있는 물질을 효율적으로 탐색하고 선별하는데 활용될 수 있다. 또한, 자신의 질환 감수성에 따라 선별적으로 식품을 섭취할 수 있는 과학적 근거자료를 제시할 수 있다는 점에서 푸드케어 서비스 산업과의 연계가 가능하다.
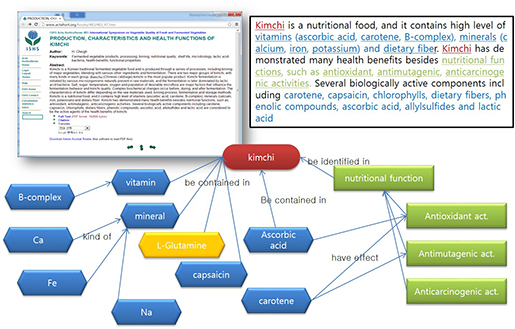
그림4. 텍스트 마이닝 기법을 이용한 김치의 생리활성 정보 수집예시 (출처:직접 작성)
식품 영양유전체 정보
많은 연구자들이 식품의 영양성분과 유전자 간의 상호작용에 대하여 관심을 두기 시작했다. 과거의 식품 영양학은 각종 영양소의 구조 및 기능을 밝히는데 초점을 맞췄다면 영양 유전체학은 개인의 유전적 특성과 상관관계가 높은 식품을 권장할 수 있는 개인별 맞춤영양학 시대로 접어들고 있다. 이러한 배경에는 사람들이 가진 유전자의 다양성에 따라 영양소 대사가 개개인의 유전적 차이에 따라 다르게 나타난다는 것이 밝혀지고 있다. Cell지에 게재된 한 논문에서는 18~70세 800명을 대상으로 같은 음식 섭취를 하게 하고 혈액 내에 glucose양을 측정한 결과에서 개인별로 glucose를 흡수하는 정도가 다르게 나타나는 것을 보고한 바 있다(그림5). 이러한 결과들은 개개인의 타고난 유전적/표현형적 특성에 따라 식품이 대사되는 정도가 다르게 나타나는 예시라 하겠다.
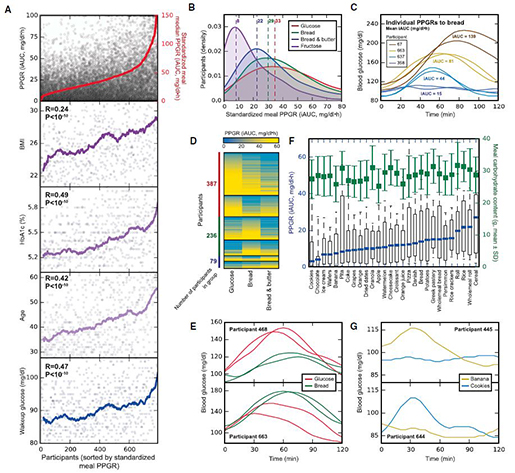
그림5. 개개인의 특성에 따른 glucose 흡수량 변화 연구 결과
(출처 : Cell, Personalized Nutrition by Prediction of Glycemic Responses 2015)
식품 영양 유전체 정보는 앞으로 유전자와 표현형 그리고 영양성분과의 상관관계가 더욱 과학적으로 규명됨으로써 양질의 정보가 될 것으로 예측하고 있다. 이처럼 축적된 정보들은 건강유지와 질병 예방을 향상할 수 있는 맞춤 의료와 식품 산업을 계속해서 가속하고 있다. 최근 habit이라는 회사는 개인 유전자 검사를 통해 자신에 맞는 식품들을 컨설팅 및 판매하는 서비스를 런칭하였으며 점차 개인 유전자 맞춤화 식품정보를 활용한 헬스케어 서비스들이 증가할 것으로 예측된다.
그림6. 식품 영양 유전체 정보와 맞춤 식품
(출처: R&D 동향, '영양 유전체학의 이해 및 연구동향' 재구성)
영양 유전체 정보를 바탕으로 맞춤형 식품 정보를 제공한 국내 사례로는 한국식품연구원과 (주)인실리코젠에서 개발한 비만 인실리코푸드시스템(http://insilicofood.co.kr)을 들 수 있다. 비만 인실리코푸드 시스템은 개인의 표현형 정보(키, 몸무게, 허리둘레, 신체활동, 컨디션등)와 유전자형 정보를 기반으로 맞춤 식품 정보를 제공하는 시스템이다. 특징적인 부분은 목표 몸무게를 설정하면 현재 표현형 정보를 기반으로 이를 달성하기 위한 식단 구성이 가능하다는 점과 개인 유전자형 정보를 입력하면 유전적으로 비만에 얼마나 위험한지 확인하고 유전자형 정보에 맞는 식품 정보를 제공한다는 점이다. 또한, 한국식품연구원 오믹스 연구결과와 식품 정보를 연결시켜 제공해주기 때문에, 과학적 근거기반의 개인 맞춤 식품 정보 시스템 구축 사례라 하겠다.
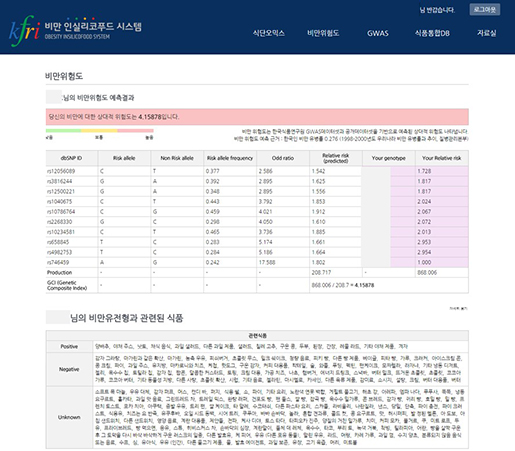
그림7. 개인 유전체 정보기반 맞춤 식품 제공 시스템
(출처 : 비만인실리코푸드 시스템 웹사이트)
식품 이력 정보
갑자기 식품 이력 정보가 왜 나오지 하고 의아해할지 모르겠지만, 필자는 식품 빅데이터가 식품 정보의 생산부터 식탁에 올라오기까지의 정보를 포괄하는 의미를 식품 빅데이터라 정의하고자 한다. 식품의 이력 정보는 식품의 생산부터 유통까지의 일련의 정보들을 의미한다. 이러한 정보들은 안심하고 먹을 수 있는 먹거리를 만드는 데 필요한 정보이다. 우리나라는 현재 정부에서 축산물에 대한 이력제 정보 시스템을 운영하고 있다. 해당 시스템을 통해 소의 출생에서부터 도축, 포장처리, 판매에 이르기까지의 정보를 확인할 수 있다. 확인방법은 축산물 상품의 이력제 번호를 모바일/웹 애플리케이션에 검색하면 이력에 대한 정보들을 확인할 수 있다. 이러한 정보들은 위생과 안전에 문제가 발생하면 그 이력을 추적하여 신속하게 대처하기 위한 유용한 정보라 할 수 있다.
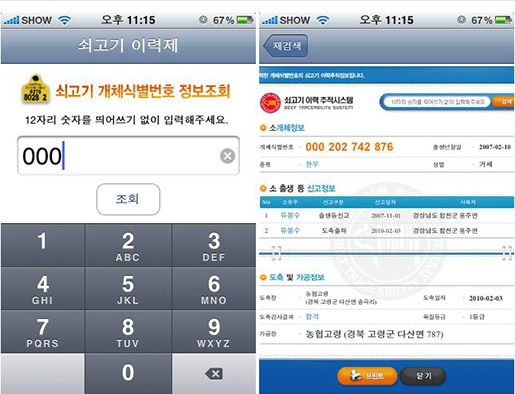
그림8. 쇠고기 이력 정보 활용예시
필자는 첫 도입 부분에 식품 데이터를 먹고사는 일이라고 표현했다. 식품 데이터는 단순한 정보의 개념에서 벗어나, 다양한 산업적 활용가치가 많은 정보기 때문이다. 중요한 것은 이렇게 많은 정보들로부터 우리는 어떤 가치를 만들어 낼 것인가이다. 식품빅데이터가 미래에 가져올 파장을 기대하며 이 글을 마무리하려고 한다.
Reference
작성자 : R&D센터 DS그룹
이상민 주임 연구원
Posted by 人Co