New 버전의 CLC Genomics Workbench를 소개합니다.
- Posted at 2013/09/30 16:46
- Filed under 제품소식
차세대 염기서열 분석 기술은 더 짧은 시간에 더 적은 비용으로 더 많은 염기서열을 결정할 수
있는 플랫폼이 계속적으로 업그레이드 되면서, 생물정보 분석은 이제 선택이 아닌 필수가 되었습니다. 또한 NGS 데이터의 분석
단계는 크게 pre-processing, assembly 그리고 assembly를 이용한 이차 분석으로 나누어 볼 수 있는데
이러한 분석 단계들을 하나의 툴에서 모두 진행하고 그 결과를 그래픽하게 확인할 수 있다면 NGS 데이터를 다루는 생물학자들이 훨씬
더 수월하게 연구를 진행할 수 있을 것입니다. 이를 위한 목적으로 개발된 CLC bio사의 CLC Genomics
Workbench는 GUI 기반의 데스크탑 솔루션으로 각 NGS 플랫폼에서 제공하는 다양한 데이터셋을 지원하여 assembly를
비롯한 이후 이차분석까지 한번에 수행할 수 있는 통합형 생물정보 분석 도구입니다.
NGS 데이터를 이용한 최근 연구들을 살펴보면 resequencing 분석에 포커싱이 맞춰지고 있으며, 이러한 연구 동향에 발맞추어 CLC Genomics Workbench의 툴들도 resequencing 분석에 초점을 맞춘 SNP, Indel detection 툴이 업그레이드되었습니다.

Variant 분석에 대한 업그레이드 내용을 이야기하기 전에 read mapping 결과를 재조정할 수 있는 툴인 ‘Local realignment’를 먼저 소개합니다. 이전에는 beta 버전으로 제공되었지만 지난 달 업그레이드가 되면서 CLC Genomics Workbench의 정식 툴이 되었습니다. Read mapping을 수행하는 과정에서 align 되지 않았던 끝 부분을 realign하여 보다 정확한 read mapping 결과를 제공해줍니다. 자, 그럼 결과를 직접 눈으로 확인하는 것이 더 신뢰가 가겠죠?

위의 그림에서 [A]는 처음 read mapping을 수행했으며, 1,2,5번째 read의
4개의 염기가 제대로 align 되지 않아 gap이 생겨난 것을 확인할 수 있습니다. [B]는 [A]의 데이터를 가지고
‘Local realignment’ 결과 화면을 보여주고 있습니다. [A]에서 gap이 발생했던 염기부분이 다시 realign된
것을 확인할 수가 있습니다. 이처럼 realign을 통해 정확한 mapping 데이터를 기반으로 이 후 variant 분석을
수행한다면 분명 고퀄리티의 결과를 얻을 수 있을 것입니다.
CLC Genomics Workbench에서의 SNP
detection은 두 가지 알고리즘으로 분석을 수행할 수 있습니다. 하나는 확률을 계산하는 ‘Probabilistic
Variant Detection’, 또 하나는 quality를 계산하는 ‘Quality-based Variant
Detection’의 분석 툴입니다. 이러한 두 가지 분석 툴 중에 데이터의 특성에 적합한 것으로 선택하여 분석한다면 더욱 정확한
잠재적인 SNP를 발굴할 수 있습니다.
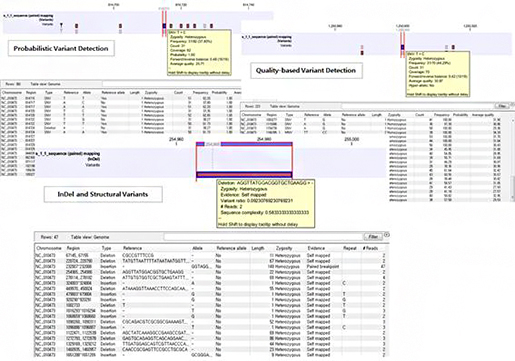
그리고 variant 분석 후 발굴된 SNP 후보들이 이미 알려진 variant 정보와 비교하여
필터링 할 수 있는 ‘Filter against known variants’와 이미 알려진 variant 정보를 추가할 수
있는 ‘Annotate from known variants’의 툴 기능도 향상되었습니다.
Read mapping을 통하여 분석된 variation들을 mapping된 서열 단위에서 그 결과를 뷰어할 수 있지만, track이라는 새로운 뷰어를 제공하여 브라우저 형태의 역할을 할 수 있도록 도와주고 있습니다. 이 또한 다양한 기능 업데이트를 통해 더 효율적으로 분석 결과를 확인할 수 있습니다.
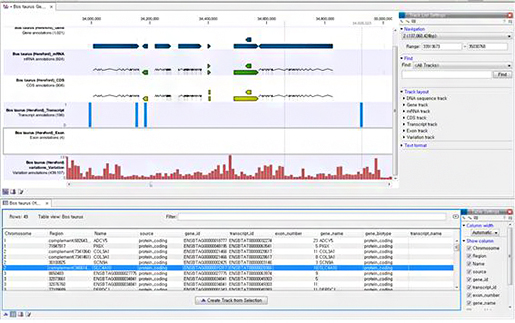
이러한 track 툴을 활용하면 위에서 보는 바와 같이 한 종의 genome 내 다양한 정보들(Gene, CDS,
Transcript, mRNA, Exon, Variation)을 한 화면에서 확인할 수 있고, 아래의 그림과 같이 Trio
analysis 툴을 이용하여 부모에게서 자녀로 유전되는 SNPs 정보나 mutation 정보, 그로 인해 나타나는 질병 등에
대한 분석 후 그 정보를 한번에 확인할 수 있습니다.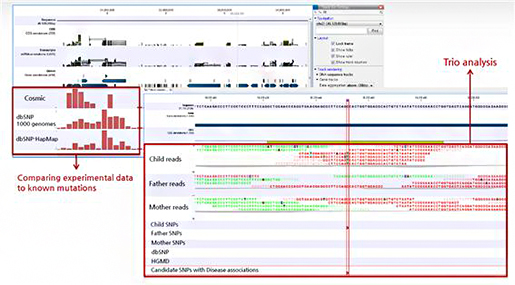
PART 2. Workflow
앞서 이야기한 variation 분석은 만약 여러
샘플에 대한 분석을 개별적으로 진행하려면, 단순한 분석을 여러번 수행함으로 시간이 많이 소요됩니다. 이렇게 여러 샘플의 데이터를
각각 동일한 분석 과정을 수행해야할 경우, workflow라는 기능을 이용하면 빠른 시간안에 쉽게 분석을 완료할 수 있습니다. 아래의 그림과 같이 분석해야하는 툴(기능)들을 선택하고, 각 툴을 순서대로 나열한 다음 각 분석단계의 output 파일을 다음 분석과정의 input 데이터로 연결만 시키면 하나의 workflow가 만들어집니다. 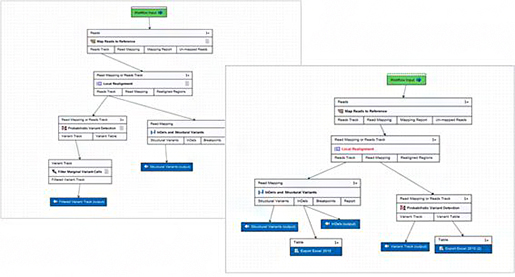
이렇게 만들어진 workflow를 이용하여 분석하고자하는 샘플 데이터만 선택해주면 일련의 과정대로 클릭 몇 번 만으로 결과
데이터를 얻을 수 있으며, workflow를 installer로 변환하여 toolbox내에 하나의 툴로써 추가할 수 있어 실험실
내에서 정규화된 분석 파이프라인을 직접 제작할 수 있습니다. 그리고 이러한 workflow를 통해 만들어지는 output 데이터를 원하는 포맷의 파일로 별도의 export가 가능합니다. 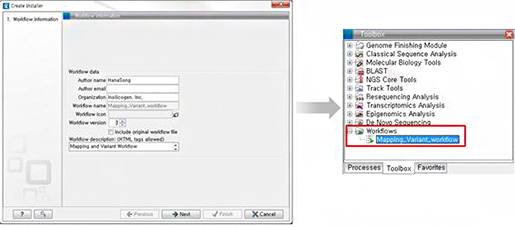
PART 3. 3D Molecule Viewing
CLC Genomics Workbench 내에는 NGS
데이터를 분석하는 툴 외에도 일반 서열 데이터를 기준으로 분석할 수 있는 다양한 생물정보 툴들이 통합되어 있습니다. 이 중
Molecule Viewer는 Protein Data Bank(PDB) database에 저장되어 있는 단백질과 다른 분자들의
structure inspection과 visualization을 수행할 수 있도록 합니다. 이
전에는 CLC Genomics Workbench를 이용하면 단백질의 3D 구조의 뷰어만 확인할 수 있었습니다. 하지만 이번
업그레이드를 통하여 3D 구조의 뷰어뿐만 아니라 서열 정보와 연동되어 3D 구조상에서 선택된 영역이 어떤 서열로 이루어져 있는지
함께 뷰어가 가능해졌습니다. Domain 및 binding site 분석하시는 연구자분들이 늘 서열과 함께 분석되어졌음 좋겠다
하셨었는데, 이젠 굉장히 유용하게 사용될 수 있겠지요?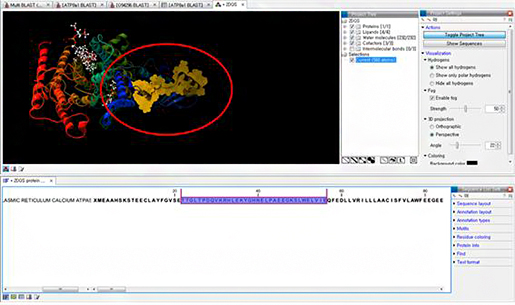
뿐만 아니라 Protein, Ligands, Water molecules, Internolecular bonds 등의 원하는 molecule 정보만을 선택하여 뷰어할 수도 있다고 합니다. 
PART 4. Phylogenetic trees
이번에 소개해드릴 기능은 계통분석을 수행하는
실험실에서는 아주 반가운 소식이 아닐까 합니다. 사실 계통분석은 굉장히 어렵고도 다양한 알고리즘이 존재하여 현재는 전문적인
소프트웨어들을 많이 사용하고 있고, CLC Genomics Workbench를 통해서는 서열을 정렬한 후 기본적인 tree까지만
제공하며 분석 결과의 수정이 어려웠던 부분이 있었습니다. 허나 이번 업그레이드를 통하여 Phylogenetic tree
module이 plug-in으로 새로 추가되어 좀 더 전문화되고, 분석 결과를 그래픽하게 확인할 수 있게 되었습니다. 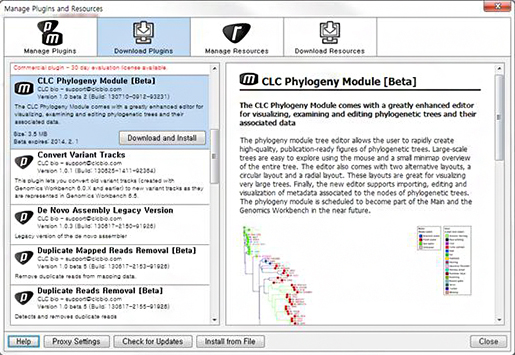
Phylogenetic tree module은 Plug-in을 통하여 다운로드 받으신 후 ‘Create Tree’ 툴로
실행합니다. 실행 과정에서는 이전 버전과 달라진 부분은 없으며 뷰어 옵션을 지정할 수 있는 Side panel을 통해 tree
결과의 뷰어 설정을 변경할 수 있습니다.
Tree layout이 아래 그림과 같이 다섯가지로 선택할 수 있어 원하는 형태의 tree구조를 figure 자료로 사용할 수 있게 되었습니다. 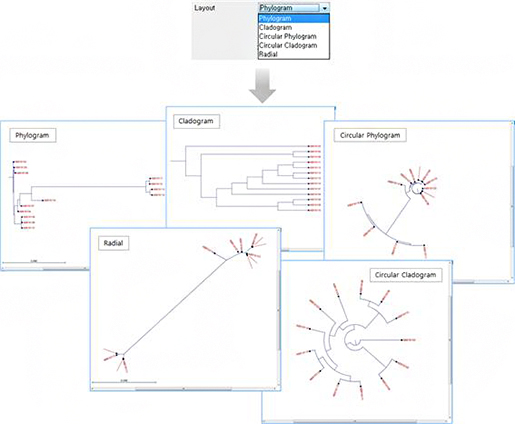
또한 이 뿐만이 아니라 메타데이터의 수정도 가능해졌습니다. 이전 버전에서 이 기능을 문의하셨던 사용자분들도 굉장히 많았었는데요, 새 버전에서는 node, label 세팅 기능의 추가로 인해 사용자가 원하는 tree 그림을 그릴 수 있게 되었습니다.
이상으로 CLC Genomics Workbench 프로그램의 주요한 업그레이드 내용을 소개해드렸습니다. 블로그를 통해 모든 정보를
알려드리기가 어려워 아쉬운점이 있지만, 소개해드린 기능 이외에도 업그레이드 및 개선된 기능들이 많이 있으니 아래의 사이트를
방문하시면 더욱 자세한 정보를 확인할 수 있습니다.
http://www.clcbio.com/products/latest-improvements/
(주)인실리코젠의 마케팅팀(Marketing@insilicogen.com)을 찾아주시기 바랍니다!
감사합니다.
작성자 : Marketing팀 송하나
Posted by 人Co
- Response
- No Trackback , No Comment
- RSS :
- https://post-blog.insilicogen.com/blog/rss/response/139
Trackback URL : 이 글에는 트랙백을 보낼 수 없습니다


