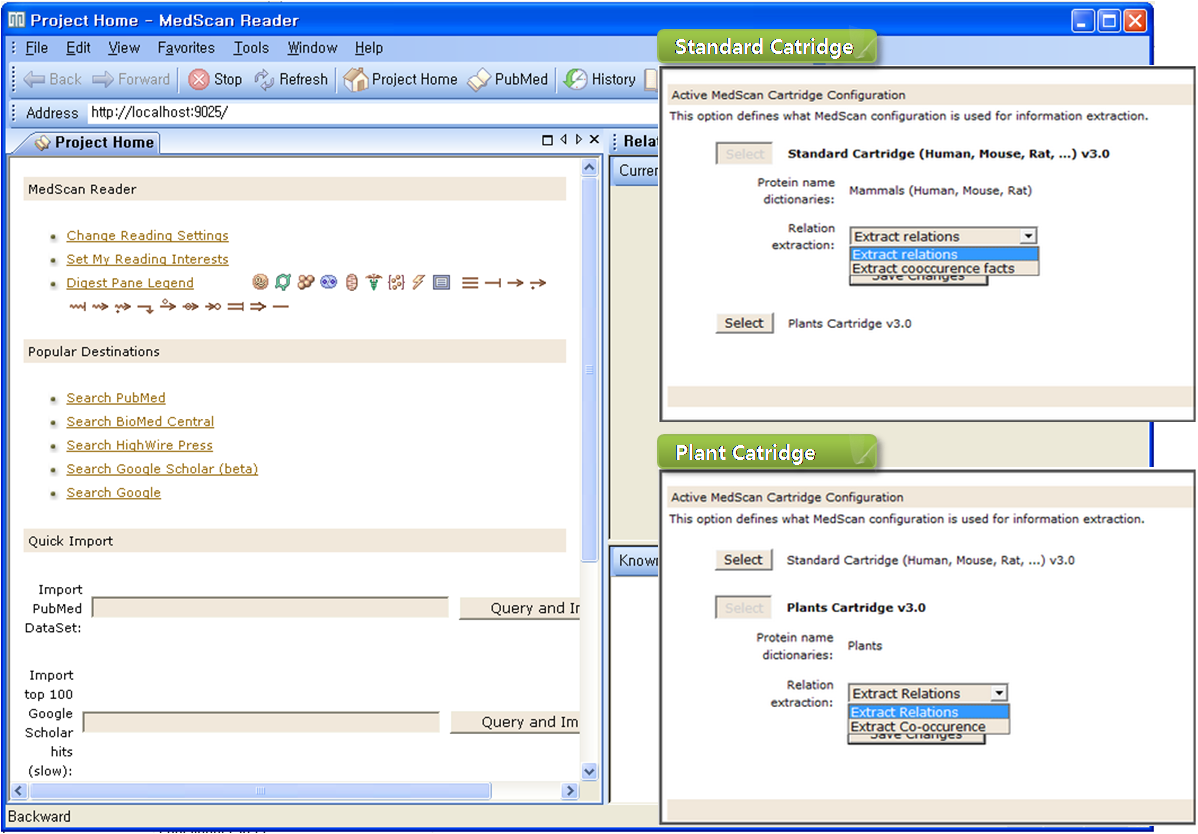
연재 순서
1. PathwayStudio 소개
2. 문헌정보를 활용한 유전자 네트워크 분석
3. Chip 실험 데이터에서의 유전자 네트워크 분석
4. Drug 발굴을 위한 지식 데이터베이스 ChemEffect
Chip 실험 데이터에서의
유전자 네트워크 분석
DNA Chip 분석 실험을 통해 유전자의 발현 양상을 분석하는 연구가 이전부터 많이 진행되어 왔다. 특정한 조건하에서 발현을
보이는 유전자가 무엇인지 검토하고 이들 유전자에 대한 다양한 정보를 검토하는 것이 이전의 연구방향이었다. 그러나
Eukaryote유전체에서는 하나의 유전자가 다양한 역할을 수행하기도 하며, 반대로 여러 개의 유전자들이 서로 상호 연관관계를
맺어서 하나의 기능을 수행하기도 한다. 따라서, 이러한 유전자들의 상호 연관관계를 도출하고자 하는 연구가 최근들어서는 중요시되고
있다. 즉 차등발현을 보이는 여러 개의 유전자들을 upstream 단계에서 조절하고 있는 요소가 무엇인지, 또한 여러 개의
유전자들이 공통적으로 타겟을 정하고 있는 질병이나, 유전자들이 무엇인지를 밝히고자 한다. 이번 블로그에서는 PathwayStudio를
이용하여 Chip 실험 데이터에 대한 유전자 네트워크 분석 방법에 대해서 알아보고자 한다.
Data importer
실험 데이터를 분석하기 위해 먼저 실험데이터를 Import 해야 한다. Pathway Studio는 실험 데이터를 매우 쉽게 입력
할 수 있도록 인터페이스가 구성되어 있다. 입력 할 수 있는 형식으로는 Gene expression, Metabolomics,
Proteomics가 있다. 입력 할 수 있는 데이터의 포맷도 아래와 같이 다양하게 제공된다.
- Tab-delemited text(Generic)
- Microsoft Excel
- GEO Datasets (GDS in SOFT format)
- Affymetrix Raw (CEL)
- Affymetrix CHP
- Agilent
- Illumina
입력할 데이터의 포맷은 DNA Chip 실험 분석을 통해서 얻어진 정보들을 탭 분리형식으로
구성되어진 파일과 엑셀 형식으로 되어 있는 파일을 불러들일 수 있다. 또한 Affymetrix, Agilent, Illumina
사와 같은 기존에 가장 많이 분석에 이용되고 있는 상용화 DNA Chip 정보 포맷도 쉽게 불러들여 분석을 진행 할 수 있다. 탭 분리형식으로 데이터가 저장된 파일을 불러들여 분석을 할 경우
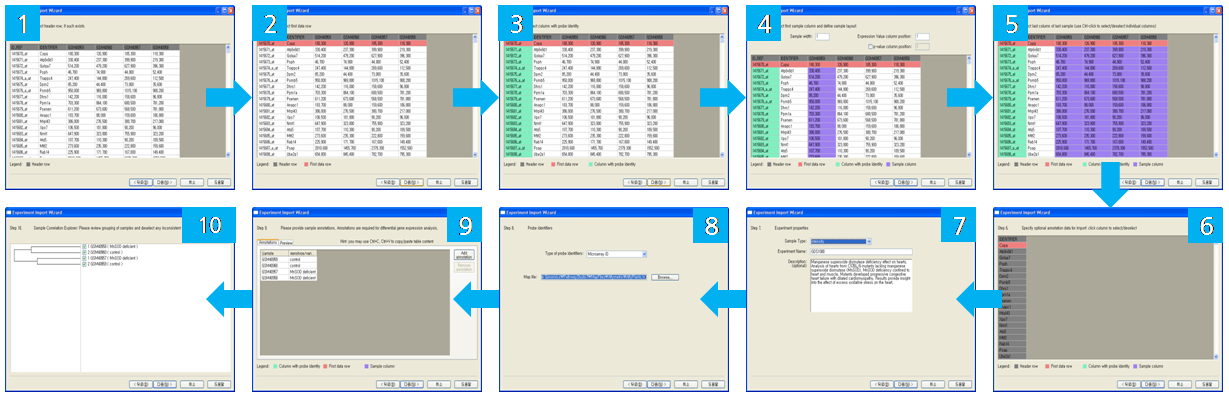
아래와 같이 모두 10가지 단계를 거치게 된다.
실험 데이터의 형식,
파일 포맷, 그리고 입력할 파일과 최종 저장할 디렉토리를 선택하면 모두 10가지 단계의 입력과정을 거치게 된다. 첫 번째 단계에서
부터 순서대로 실험 데이터의 헤더 설정, 데이터가 시작되는 행의 지정, Probe identity를 표현하는 컬럼 지정, 샘플의
layout 설정, 데이터의 마지막 컬럼 지정, 부가적으로 사용될 annotation으로 컬럼 설정, 샘플 타입, 부가적으로
Probe를 식별하는데 사용하는 Identifier, expression 분석에 사용되는 annotation 컬럼을 선택한다.
마지막 열 번째 단계에서는 반복실험을 수행한 샘플들간의 상호 연관성을 볼 수 있는 sample correlation 단계로 샘플
간에 가까운 상관관계가 있는 것끼리 그룹으로 설정하여 Tree 형태로 보여준다. 여기에서 correlation이 잘못된 경우
분석에서 제외할 샘플을 선별 할 수 있다.
그림 1. Chip 실험 데이터 import stepResult and
displays
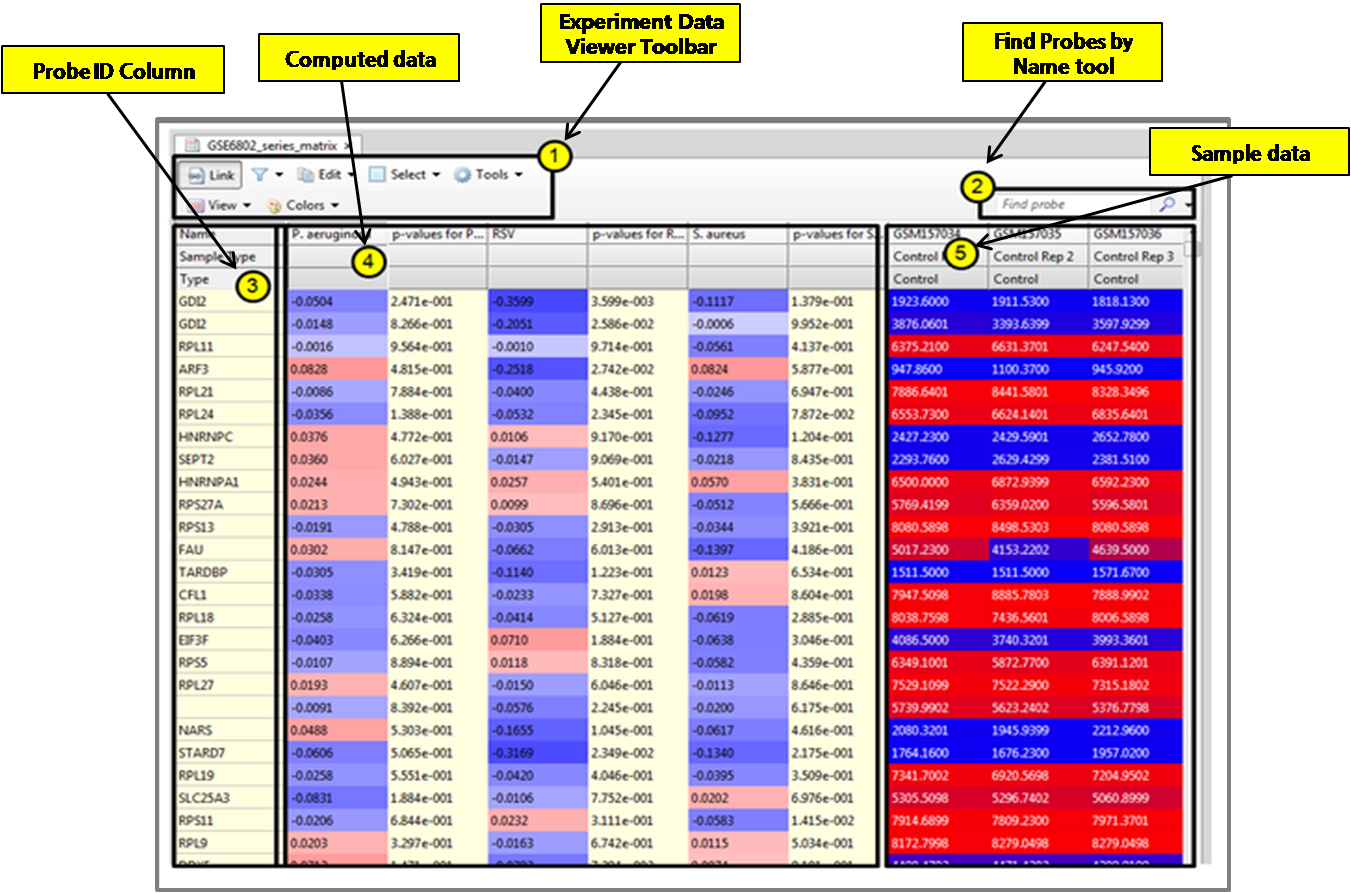
모든 입력 단계가 완료되면 아래와 같이 Pathway Studio에 실험데이터가 입력 된 것을 확인할 수 있다. 실험 데이터
뷰어는 크게 5가지 부분으로 나뉜다.
- 데이터를 분석하거나 편집할 때 많이 사용하는 기능을 모아 놓은 Toolbar
- Probe를 빠르게 검색하여 찾을 수 있는 검색창
- Probe ID 컬럼
- 발현 차이를 보기 위해 t-test 통계 수치가 계산된 컬럼
- 샘플 데이터
그림 2. 실험 데이터 Viewer 인터페이스
Data analysis
algorithms
Pathway Studio에서 실험데이터를 분석하는 알고리즘은 크게 세 가지가 있다.
- Fisher's Exact Test
- Gene Set Enrichment Analysis
- Sub-Network Enrichment
Fisher's Exact Test와 Gene Set Enrichment Analysis는
Enrichment를 분석하는 서로 다른 알고리즘이고 Sub-Network Enrichment는 ResNet에서 사용자가 직접 Gene
Set을 설정해 주는 것으로 알려진 Gene Set을 사용하는 앞의 두 알고리즘과는 다르다.
먼저 Fisher's Exact Test를 수행하기 전에 통계적으로 유의한 유전자 리스트를 확인하기
위해 필터링 과정을 거친다. Toolbar에서 Filter Probes by value를 클릭하면 대화창이 나타난다. 필터링을
적용하고자 하는 샘플을 선택하고 Probe 범위와 P-value cutoff 값을 입력하여 필터링을 수행 한다. 이제
Filter를 통해 나온 데이터들을 이용해서 Fisher's Exact Test를 수행한다. 분석하고자 하는 데이터 컬럼에서
마우스 오른쪽을 눌러 Find Pathway/Groups Enriched with Selected Entities를 선택한다.
대화창이 나타나는데 여기에서 실험 데이터를 대상으로 Fisher's Exact Test를 수행 할 Ontology나
Pathway를 선택한다.
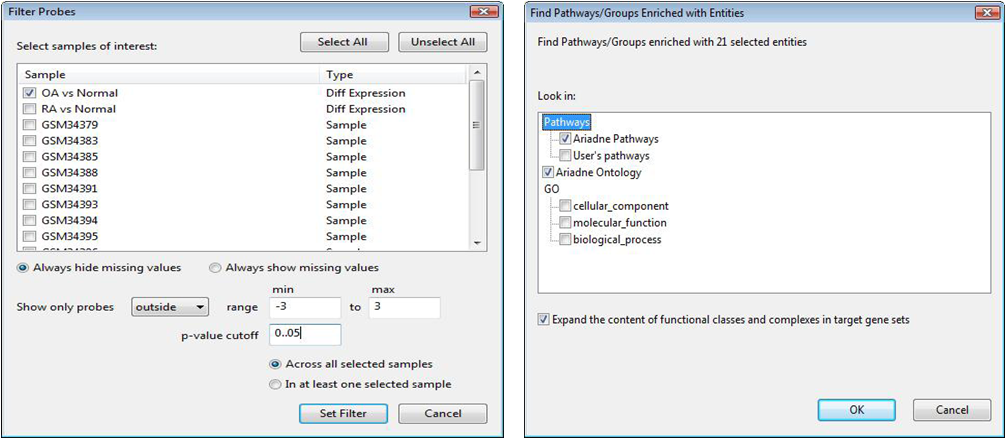
그림 3. Fisher's Exact Test 분석 1) Filter Probes by value
2) Find Pathway/Groups Enriched with Selected Entities
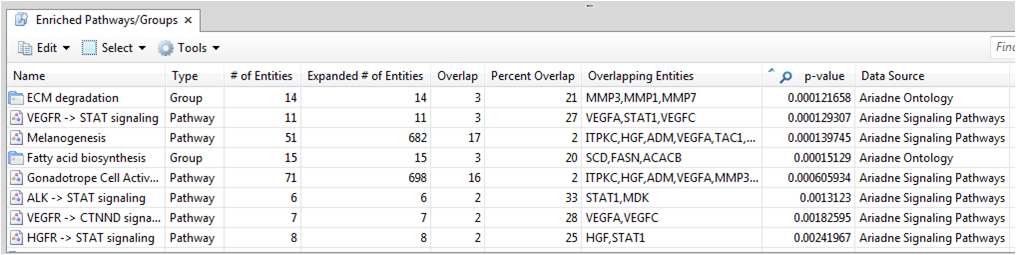
분석이 완료되면 하단에 P-value를 기준으로 정렬된 분석 결과가 테이블 형태로 나타난다. 결과 테이블에서는 분석할 때 선택한
샘플 유전자들이 어떤 Pathway나 Group에 가장 가깝게 해당되는지 알 수 있다. 아래의 결과 테이블을 예로 들어보면
Fisher's Exact Test를 할 때 선택해준 샘플 데이터가 결과 테이블 첫 번째 행에 있는 ECM degradation
즉, ECM 단백질의 분해에 관여하는 Group에 속한다는 것을 알 수 있다. ECM degradation group에는 14개의
Entity가 있는데 그 중에서 선택한 샘플 데이터와 중복이 되는 것은 3개이고 구체적으로 어떤 유전자가 중복이 되는지
리스트도 함께 보여준다.
그림 4. Fisher's Exact Test 분석 결과 테이블
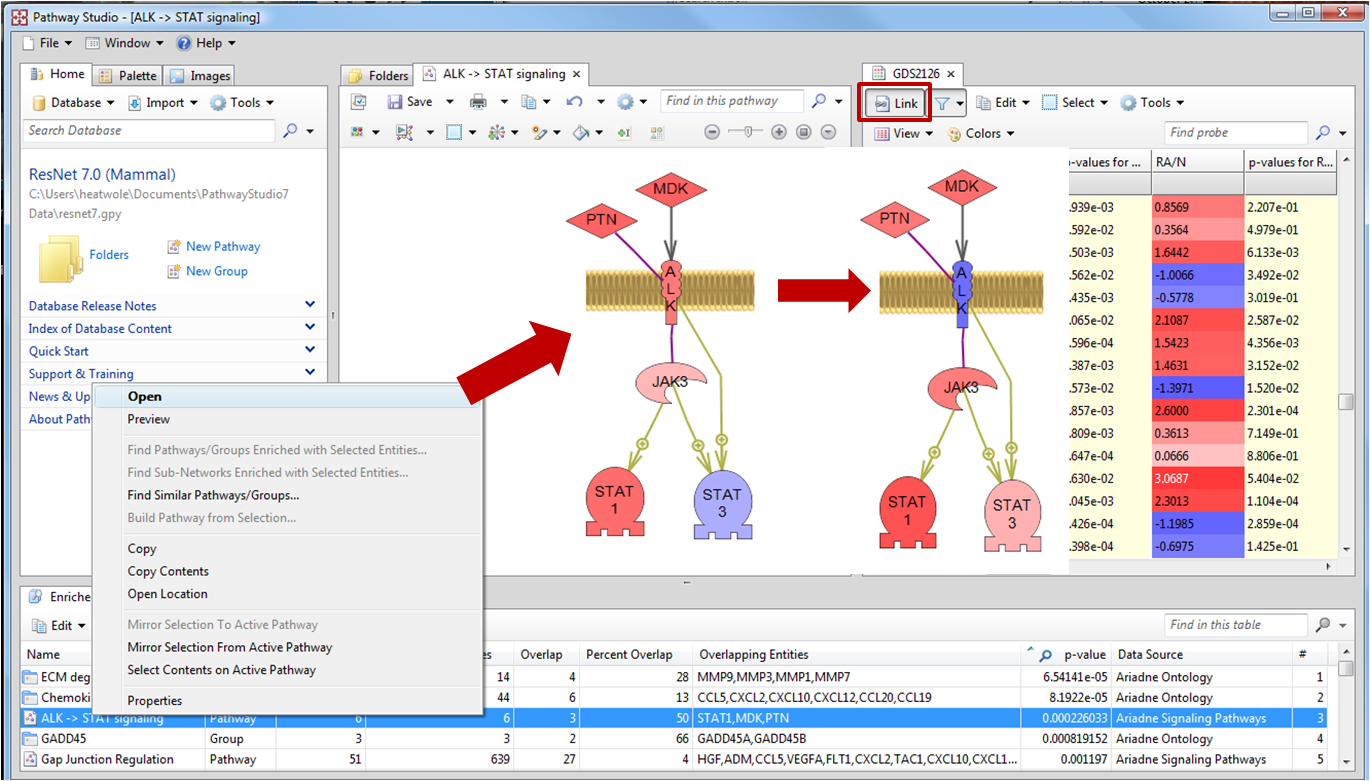
결과 테이블에서 보고자 하는 Pathway를 오픈하면 Graph view에서 볼 수 있다. 이 Pathway에 있는
Entity들이 입력한 실험 데이터에 있는 유전자들 중에서도 발현이 되고 있는지 보고자 한다면 실험 데이터 viewer의
Toolbar에서 Link 버튼을 이용한다. 발현이 되고 있다면 Entity의 색이 변하는 것을 볼 수 있다.
그림 5. Fisher's Exact Test 분석 결과 Pathway 보기 Link 메뉴를 통해
분석결과 나온 Pathway와 실험 데이터의 유전자들이 서로 link 되어 있는지 확인할 수 있다.
두 번째 알고리즘인 Gene Set enrichment 분석은 Fisher's Exact
Test와 비슷한 분석이지만 실험 데이터에 서열화 방법을 적용하였다는 차이점이 있다. Gene Set enrichment 분석을 할
때에는 Filter를 적용하지 않고 분석을 시작한다. Enrichment 분석 할 알고리즘으로 2가지가 제공되는데
Mann-Whitley Utest와 Kolmogorov-Smirnov가 그것이다. 두 가지 모두 순위척도 자료를 위한 비모수
통계방법으로 두 모집단 사이에 유의한 차이가 있는지를 검정할 때 사용한다.
Gene Set enrichment 분석도 마찬가지로 분석이 완료되면 그림 3과 같은 결과 테이블을
제공한다. 결과 테이블에서 Fisher's Exact Test와 다른점이 있다면 Median fold change 값을 제공한다는
것이다. 이 값을 통해 측정된 Entity 그룹에 대한 fold change 값의 분포를 알 수 있고, 결과 set에서 더 up
regulated 되거나 더 down regulated 되는 그룹을 빠르게 확인 할 수 있도록 정보를 제공해 주고 있다.
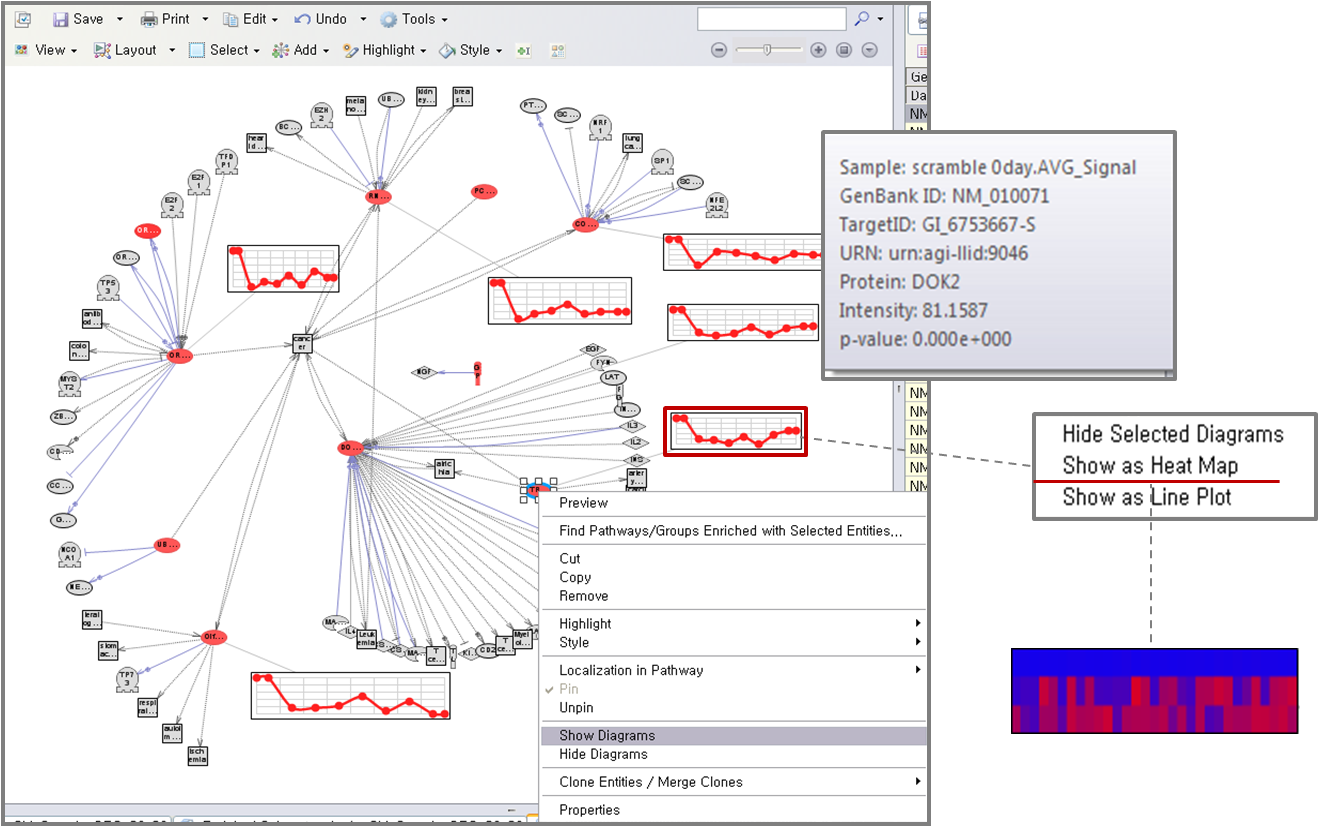
이렇게 분석 결과 나온 pathway는 여러 가지 pathway를 합쳐서 보거나, 공통된 것 또는
공통된 것을 제외한 나머지 부분만을 볼 수도 있다. 또한 실험데이터가 Time-course로 진행된 실험이라면 특정 Entity가
시간에 따라 어떻게 발현이 달라지는지 볼 수 있다. 보고자 하는 Entity를 하나 선택하고 마우스 오른쪽을 클릭하면 Show
diagram이 있다. 이것을 클릭하면 그림에서 보는 것과 같이 Line plot 형태로 그려진 diagram이 생성된다. 다시 이
다이어그램을 클릭하고 마우스 오른쪽을 노르면 Show as Heat Map 메뉴가 있는데 이것은 Line plot 형태의
다이어그램을 Heat Map 형태로 바꾸어 볼 수 있는 역할을 한다.
그림 6. Visualizing time-course data on pathway Pathway Studio를 통해서 실험데이터 차등발현유전자를 선별하고 여러가지 통계분석을 통해 이 유전자들을 조절하는
상위조절인자를 분석하거나 차등발현유전자들이 공통적으로 작용하고 있는 질병, 세포내 프로세스 등을 알아볼 수 있었다. 앞선 연재에서
설명한 Pathway Studio의 ResNet
DB 또는 문헌정보를 활용한 유전자 네트워크 정보와도 비교 분석하면서 데이터를 해석하면 좀 더 의미있는 결과가 도출될 것이다.
Posted by 人Co
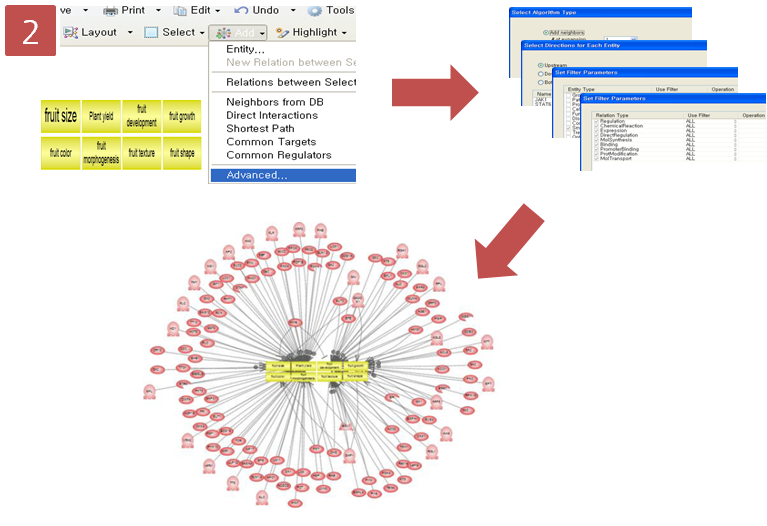 Step 3. Entity Table 보기(View > Entity Table View)
Step 3. Entity Table 보기(View > Entity Table View)
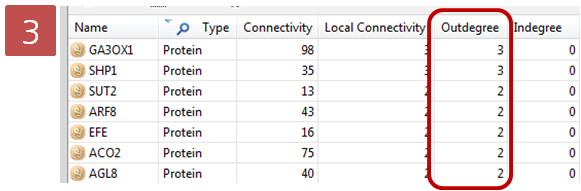
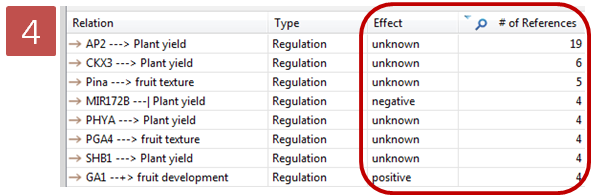 Step 5. Active Style 변경
Step 5. Active Style 변경