연재 순서 1. Assembly 2. Variation study 3. Expression study
4. Epigenomics
5. Genome Annotation
6. Next Generation Bioinformatics
7. Data Management for web 2.0 Era
8. Semantic Network for Integrated Biology Data
9. Gene Network Discovery by Text-mining
10. Centralization for High-throughput Data Analysis
Quipu Issue Paper 기술 소식지 첫 번째 연재로 NGS Assembly 중에
Reference assenbly에 대해 알아보도록 하겟습니다.
1. Next Generation Sequencing?
1-2. Assembly
Next Generation Sequencing(NGS)으로 인한 무제한적인 서열 데이터 생산은 이후 생물정보학적 분석의 가장 큰 도전 과제가 되었다. 일차적으로 많은 양의 데이터 관리부터 분석과정 마다의 computing 속도가 문제로 제기 되었다. 그중 가장 첫 번째 단계가assembly이다. NGS 서열의 assembly는 그 목적에 따라 크게 reference assembly와 de novo assembly로 구분 지어진다. Reference assembly의 경우 variation 및 epigenetics 연구에 주로 이용되고 de novo assembly의 경우 기존의 genome project에서 진행하던 whole genome sequencing에 이용되고 있다. 세부적인 내용을 다음에서 알아보자.
1-2-1. Reference assembly
Re-sequencing을 통한 기존의 reference 서열과의 비교로 유전체 상의 variation 연구를 목적으로 진행하는 시퀀싱은 주로 single reads를 얻는 시퀀싱 보다는 paired-end 시퀀싱이 수행된다. 그 이유는 다양한 질병 관련 유전자의 SNP 및 CNV 분석을 위해서는 single reads 보다는 paired-end reads가 더 유용하기 때문이며, 이들 데이터는 앞서 언급한 다양한 플랫폼에서 생산되고 있다. 이렇게 생산된
NGS 데이터를 분석할 수 있는 프로그램은 오픈 소스로 제공 되는 것과 그렇지 않은 것들로 여러 개가 존재한다. 그 중 오픈 소스로 제공하는 SOAP[1], MAQ[2] 그리고 ZOOM[3]은 paired-end short read에 최적화 되어 있고, Newbler는 long reads인 454 reads에 최적화 되어 있다. 이렇게 대부분 특정
NGS 플랫폼에서 생산된 데이터만을 다룰 수 있도록 고정화되어 있는 것에 반해 CLC bio사의 CLC NGS Cell[4]은 언급된 모든 플랫폼의 데이터를 분석할 수 있는 장점이 있다[14]. 이들 프로그램에 대하여 좀 더 자세히 알아보자.
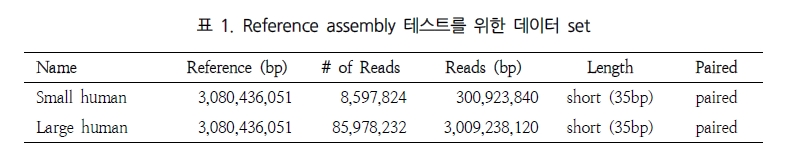
NGS assembly 프로그램을 평가하는데 있어 가장 큰 이슈는 분석 속도와 결과의 정확성, 그리고 그 외 분석의 용이성을 들 수 있다. 이들에 대한 비교 분석을 위해 표 1에서 보여 지는 paired-end의 short reads을 대상으로 여러 가지 분석을 수행하였다. 이러한 분석은 64-bit Xeon E5420 CPUs에 32 GB memory system에서 수행되었다[1].
첫 번째인 분석 속도에서는 CLC NGS Cell이 가장 빠른 것으로 평가 되었다(표 2)[5].
SIMD 기술을 이용한 병렬 데이터 처리로 속도 면에서 월등히 높은 성능을 나타내었다. 그 외 SOAP의 경우 reference 서열을 2-bit로 전환하여 index 파일을 이용한 연산 처리로 좋은 결과를 보이고 있다(2009.11 현재 SOAP의 경우 업그레이드를 통해 분석 속도가 많이 향상 되었다).
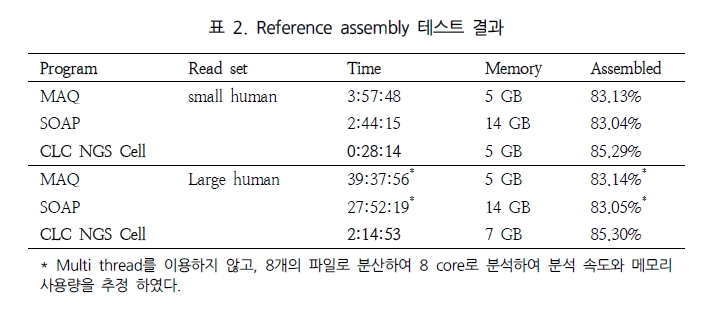
특히, Maq의 경우 Illumina와 SOLiD의 paired-end reads를 대상으로 human 유전체에 맵핑할 경우 CPU time으로 10 시간 동안 백만 개 paired-end reads를 assembly 할 수 있다고 밝혔다[2]. 같은 시험을 위해 자체적으로 SOLiD reads를 대상으로 CLC NGS Cell을 이용하여 분석했을 때 CPU time으로 5시간 28분에 분석이 완료됨을 확인하였다. 두 번째로 NGS read의 alignment 비율 및 정확성을 살펴보았다. 최근 논문 PLoS ONE에 기재된 ‘Mapping Accuracy of Short Reads from Massively Parallel Sequencing and the Implications for Quantitative expression Profiling’에서는 BLAT[15], SSAHA2[16], Bowtie[17], SeqMap[18], MAQ, CLC NGS Cell을 대상으로 다양한 종의 데이터로 프로그램의 정확성을 다각도로 분석한 결과를 발표 하였다[6]. 그 결과 그림 1에서 보여 지는 것과 같이 SSAHA2와 CLC NGS Cell이 높게 평가되었다. 이 중 SSAHA2는 Sanger institute에서 개발된 프로그램으로 현재 SOLiD data를 제외한 모든 플랫폼의 데이터를 분석할 수 있다[7]. 기본적으로 Smith-Waterman alignment를 수행하며 2-bit로 전환하여 정확한 assembly를 수행한다. 그 다음 CLC NGS Cell은 모든 플랫폼의 데이터를 처리함과 동시에 SSAHA2와 같이 안정적으로 reads 길이에 관계없이 정확한 assembly를 수행하고 있다. 또한 특이할만한 점은 yeast, drosophila, arabidopsis 그리고 human을 대상으로 한 다양한 데이터로 short reads와 long reads(>50bp)에 대한 프로그램 성능을 비교 하였음에도 불구하고(MAQ: short read만이 분석 가능), 프로그램별로 일관성 있는 결과를 보여주고 있다는 것이다. 각기 다른 종과 read 길이로 약간의 차이는 보이나 전반적으로 동일한 분석 패턴을 보이고 있어, 이는 곧 데이터의 특성보다는 프로그램별 알고리즘의 차이가 분석 결과에 더 많은 영향을 미치는 것으로 해석된다. 따라서
NGS를 이용한 분석에서 다양한 프로그램을 이용하여 분석 파이프라인을 구축하는 것 보다는 사전에 충분한 테스트를 통해 동일한 알고리즘으로 구성된 프로그램을 이용하는 것이 결과의 안정성과 정확성을 높일 수 있는 하나의 방법이 될 수 있겠다.
NGS를 이용한 연구에서 특히 re-sequencing을 하는 경우 대부분 유전체 상의 variation 연구를 목적으로 진행된다. 따라서 re-sequencing된 데이터는 기존의 reference 서열과는 다른 variation을 가지는 특성이 있으므로 이를 고려한 assembly 알고리즘이 필요하다.
 그림 1. 프로그램별 다양한 데이터 셑으로 구성된 reference assembly 시험 결과. 회색바는 alignment 된 비율, 붉은색바는 부정확한 alignment를 각각 나타낸다 | 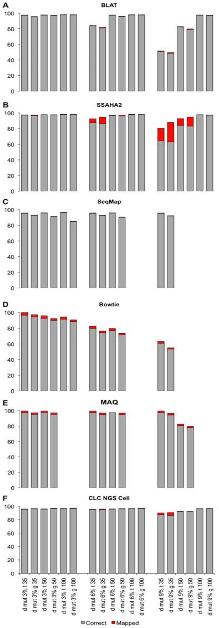 그림 2. Reads의 다양한 mutation 비율에 따른 mapping의 정확성 시험. Drosophila genome과 transcripts를 reference로 하여 reads의 mutation 비율을 각각 3%, 6%, 9%로 조정하여 mappping을 수행. 회색바는 alignment된 reads의 비율을 의미하며 붉은색 바는 부정확하게 alignment된 비율을 나타낸다. |
그림 2에서는 각 프로그램별 variation을 고려한 assembly 결과를 보여주고 있다[6]. Drosophila의 transcripts와 유전체 서열을 각각 reference로 하고 mutation 비율이 각기 다른 NGS reads를 맵핑하여 프로그램의 정확성을 확인 하였다. 이도 역시 CLC NGS Cell과 SSAHA2가 가장 우수한 결과를 보이고 있다. 그러나 CLC NGS Cell의 경우 mutation 비율에 상관없이 안정적인 정확성을 보이고 있는 반면, SSAHA2는 mutation 비율이 커짐에 따라 정확성이 떨어지는 문제점을 들어내고 있다. 따라서 SSAHA2를 이용할 경우 사전에 데이터의 특성을 미리 파악하여 적절히 이용하는 것이 좋을 듯하다.
마지막으로 분석의 용이성을 여러 가지 측면으로 살펴보았다.
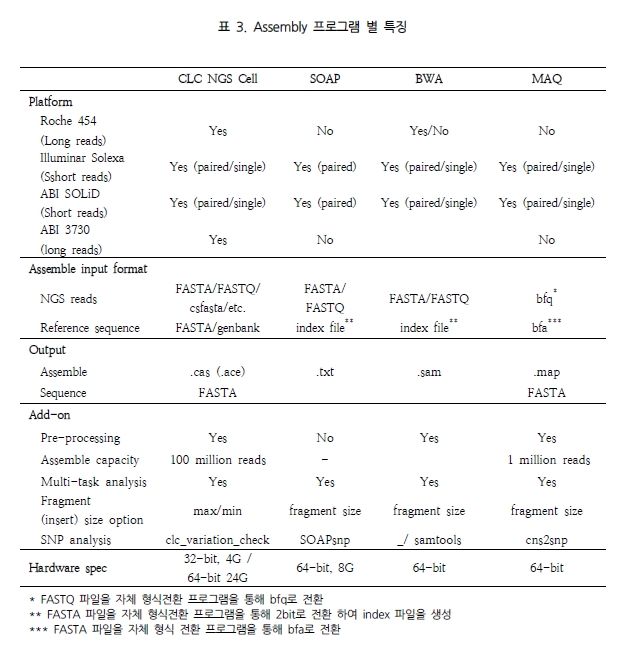
NGS 분석을 목적으로 개발된 MAQ, SOAP, 그리고 CLC NGS Cell은 모두 웹에서 다운로드가 가능하다. 이 중 CLC NGS Cell은 압축만 해제하면 바로 실행할 수 있는 바이너리 파일을 제공하고 있고, SOAP과 MAQ은 각각 압축 해제 후 compile을 통해 쉽게 설치가 가능하다.
이 후 분석에 필요한 입력 데이터 형식은 CLC NGS Cell이 가장 호환성이 좋아 FASTA, FASTQ, csfasta(SOLiD), Scarf, Sff의 모든 형식의 파일을 입력 받을 수 있었으며 SOAP과 MAQ은 각각 프로그램에 맞는 형식이 따로 존재하여, 이들 형식으로 전환할 수 있는 프로그램을 따로 제공하고 있는 실정이다. 이때 paired-end reads의 경우 분석 결과의 신뢰성과 정확성을 높이기 위해 assembly 수행 전에 서열이 쌍으로 존재하는지 여부를 체크하게 되는데, 이를 점검할 수 있는 프로그램을 CLC NGS Cell과 MAQ은 제공하고 있다. 이는 분석자에게 NGS reads의 전처리 과정을 수월하게 진행할 수 있게 하는 편의성도 고려된 것이다.
Reference 서열 또한 CLC NGS Cell은 FASTA 형식과 genbank 형식의 파일을 바로 입력 받을 수 있는 장점을 가지고 있으며, 나머지 프로그램은 각각의 형식으로 전환할 프로그램을 제공하여 한 번의 분석 단계를 더 수행하도록 되어있다. 그 외 분석에 필요한 옵션사항은 약간의 차이를 보일뿐 큰 차이는 없었으나, 다음 분석을 위한 assembly 결과 파일의 데이터 호환성에서는 CLC NGS Cell과 MAQ이 SOAP보다는 우위를 나타내었다. 마지막으로 NGS 분석 프로그램에서 중요하게 체크해야 할 사항 중에 하나는 assembly 과정을 나눠 진행하고 이후에 결과를 하나로 합쳐 볼 수 있는 기능이 있는지를 살펴보는 것이다.
제한된 computing power로 이처럼 큰 사이즈의 유전체 서열과 NGS reads를 분석해야 하므로 한 번에 데이터를 분석 한다는 것은 매우 어려운 일이다. 따라서 가능한 분산 처리로 데이터를 나눠 분석하고 이들을 통합할 수 있는 기능이 있어야만 한다. 다행히 이러한 기능은 CLC NGS Cell(join_assemblies)과 MAQ(mapmerge)에서 제공을 하고 있었다. 이들 각각의 특징은 표 3에서 자세히 확인할 수 있다.
다음 연재에서는 Reference assembly에 이어서 NGS Assembly 중에
de novo assembly에 대해 알아보도록 하겠습니다. 많은 관심 부탁드립니다.
참고문헌 1. Li R, Li Y, Kristiansen K, Wang J. (2008) SOAP: short oligonucleotide alignment program. Bioinformatics 24, 713–714 (http://soap.genomics.org.cn/index.html)
2. Li H, Ruan J, Durbin R. (2008) Mapping short DNA sequencing reads and calling variants using mapping quality scores. Genome Res 18, 1851–1858 (http://maq.sourceforge.net/index.shtml)
3. Lin H, Zhang Z, Zhang MQ, Ma B, Li M. (2008) ZOOM! Zillions of oligos mapped. Bioinformatics 24, 2431–2437 (http://www.bioinfor.com)
4. CLC NGS Cell : http://www.clcbio.com
5. White paper on reference assembly on the CLC NGS Cell 2.0 (www.clcbio.com)
6. Palmieri N, Schlötterer C. (2009) Mapping accuracy of short reads from massively parallel sequencing and the implications for quantitative expression profiling. PLoS One. 28, 4(7):e6323.
7. Ning Z, Cox AJ, Mullikin JC. (2001) SSAHA: a fast search method for large DNA databases. Genome Res. 10, 1725-1729.
8. Roche 454 : http://www.454.com/
9. Zerbino DR, Birney E. (2008) Velvet: algorithms for de novo short read assembly using de Bruijn graphs. Genome Res 18, 821–829.(http://www.ebi.ac.uk/~zerbino/velvet/)
10. Newbler : 454 bundle program
11. Birol I, Jackman SD, Nielsen CB, Qian JQ, Varhol R, Stazyk G, Morin RD, Zhao Y, Hirst M, Schein JE, Horsman DE, Connors JM, Gascoyne RD, Marra MA, Jones SJ. (2009) De novo transcriptome assembly with ABySS. Bioinformatics. 21, 2872-2877
12. White paper on de novo assembly in CLC NGS Cell 3.0 beta (www.clcbio.com)
13. Andreas T., Eva T., Thomas B., Alexander G., Ulrike L. and Alfred P. Ultrafast de novo sequencing of the human pathogen Corynebacterium urealyticum with the Genome Sequencer System (http://www.454.com/downloads/protocols/Whole_Genome_Sequencing_And_Assembly.pdf)
14. Horner DS, Pavesi G, Castrignanò T, De Meo PD, Liuni S, Sammeth M, Picardi E, Pesole G. (2009) Bioinformatics approaches for genomics and post genomics applications of next-generation sequencing. Brief Bioinform. [Epub ahead of print]
15. Kent WJ. (2002) BLAT--the BLAST-like alignment tool. Genome Res. 4, 656-664.
16. Ning Z, Cox AJ, Mullikin JC. (2001) SSAHA: a fast search method for large DNA databases. Genome Res. 10, 1725-1729.
17. Langmead B, Trapnell C, Pop M, Salzburg SL. (2009) Ultrafast and memory-efficient alignment of short DNA sequences to the human genome. Genome Biology. 3, R25
18. Jiang H, Wong WH. (2008) SeqMap: mapping massive amount of oligonucleotides to the genome. Bioinformatics. 20, 395-396.
Posted by 人Co
 이번 교육은 당사에서 발간한 Quipu Issue Paper 2호의 "NGS 시대의 분석전략 2"을 중심으로 최근 가장 이슈가
되고 있는 NGS 데이터의 assembly, 그리고 그 이후에 진행할 수 있는 다양한 분석들에 대한 내용들을 크게 3가지
세션으로 나누어 구성하였습니다. 또한 생물정보 분야의 중심 역할을 하고 있는 한국생명공학연구원
국가생물자원정보관리센터(KOBIC)의 많은 연구원분들을 대상으로 진행되었습니다.
이번 교육은 당사에서 발간한 Quipu Issue Paper 2호의 "NGS 시대의 분석전략 2"을 중심으로 최근 가장 이슈가
되고 있는 NGS 데이터의 assembly, 그리고 그 이후에 진행할 수 있는 다양한 분석들에 대한 내용들을 크게 3가지
세션으로 나누어 구성하였습니다. 또한 생물정보 분야의 중심 역할을 하고 있는 한국생명공학연구원
국가생물자원정보관리센터(KOBIC)의 많은 연구원분들을 대상으로 진행되었습니다. NGS 데이터의 assembly는 유전체 분석에 있어서 데이터 플랫폼의 종류와 어떤 어셈블러를 사용하느냐에 따른 분석 전략 및
파이프라인은 꼭 필요할 것이라 생각합니다. 이에 첫 번째 세션은 De novo assembly와 Reference
assembly에 사용되고 있는 여러 가지 어셈블러들의 종류, 장단점 비교, 실제 데이터 벤치마킹 결과 등에 대한 내용으로
준비하였고, 발표 중간중간 관련 사항에 대한 질문과 열띤 토론으로 참석하신 연구원분들의 많은 관심을 받았습니다.
NGS 데이터의 assembly는 유전체 분석에 있어서 데이터 플랫폼의 종류와 어떤 어셈블러를 사용하느냐에 따른 분석 전략 및
파이프라인은 꼭 필요할 것이라 생각합니다. 이에 첫 번째 세션은 De novo assembly와 Reference
assembly에 사용되고 있는 여러 가지 어셈블러들의 종류, 장단점 비교, 실제 데이터 벤치마킹 결과 등에 대한 내용으로
준비하였고, 발표 중간중간 관련 사항에 대한 질문과 열띤 토론으로 참석하신 연구원분들의 많은 관심을 받았습니다. 두번째 세션은 SNP 분석 방법 및 최근 capture array 분석의 실제 연구사례, 관련 솔루션 등을 소개한
variation 분석 파트와 EST 데이터를 이용한 functional annotation, Organism-specific
분석, Ortholog/Paralog 유전자 분석방법 등에 대한 expression 분석 파트로 구분되어 진행되었으며 마지막
세션은 NGS와 생물정보 파이프라인을 이용한 Genome annotation에 대한 내용으로 현재 NGS 염기서열 결정 이후
문제점 및 이슈를 분석하고 효율적인 전략들을 소개하였습니다. 또한 structural annotation과 functional
annotation의 분석 방법 및 실제 Codes팀의 분석 컨설팅 파이프라인 관련하여도 설명 드릴 수 있는 좋은시간이
되었습니다.
두번째 세션은 SNP 분석 방법 및 최근 capture array 분석의 실제 연구사례, 관련 솔루션 등을 소개한
variation 분석 파트와 EST 데이터를 이용한 functional annotation, Organism-specific
분석, Ortholog/Paralog 유전자 분석방법 등에 대한 expression 분석 파트로 구분되어 진행되었으며 마지막
세션은 NGS와 생물정보 파이프라인을 이용한 Genome annotation에 대한 내용으로 현재 NGS 염기서열 결정 이후
문제점 및 이슈를 분석하고 효율적인 전략들을 소개하였습니다. 또한 structural annotation과 functional
annotation의 분석 방법 및 실제 Codes팀의 분석 컨설팅 파이프라인 관련하여도 설명 드릴 수 있는 좋은시간이
되었습니다. 이렇게 바쁜 와중에도 하루의 일정을 직접 방문하여 소화해주신 KOBIC 연구원분들께 감사의 인사를 드리며, 진행된 교육으로
인해서 NGS 데이터를 분석하고 연구하시는데 조금이나마 도움이 되었으면 하는 바램입니다. 또한 "NGS시대의 분석전략 3"의
발간도 부탁하실 정도로 기술소식지와 세미나에 큰 관심을 보여주셔서 더욱 뜻 깊은 시간이었고, 앞으로도 이러한 교육의 자리를 많이
준비하도록 노력하겠습니다.
이렇게 바쁜 와중에도 하루의 일정을 직접 방문하여 소화해주신 KOBIC 연구원분들께 감사의 인사를 드리며, 진행된 교육으로
인해서 NGS 데이터를 분석하고 연구하시는데 조금이나마 도움이 되었으면 하는 바램입니다. 또한 "NGS시대의 분석전략 3"의
발간도 부탁하실 정도로 기술소식지와 세미나에 큰 관심을 보여주셔서 더욱 뜻 깊은 시간이었고, 앞으로도 이러한 교육의 자리를 많이
준비하도록 노력하겠습니다. 책자로 발간되었지만, 이번 세미나 내용을 포함한 NGS시대의 분석전략은 더욱 많은 연구자분들께 유익한 정보를 제공해 드리고자 블로그 연재도 계속 진행중입니다. 이와 관련한 자세한 문의사항은 저희 (주)인실리코젠의 Codes팀에게 연락 부탁드립니다.
책자로 발간되었지만, 이번 세미나 내용을 포함한 NGS시대의 분석전략은 더욱 많은 연구자분들께 유익한 정보를 제공해 드리고자 블로그 연재도 계속 진행중입니다. 이와 관련한 자세한 문의사항은 저희 (주)인실리코젠의 Codes팀에게 연락 부탁드립니다.