- [릴리즈] QIAGEN OmicSoft Lands API 공유
- [릴리즈] QIAGEN OmicSoft Lands API 링크드인 공유 페이스북 공유 트위터 공유
-
Latest Improvements for QIAGEN OmicSoft Software
QIAGEN OmicSoft Lands API는 선별된 오믹스 데이터에 대한 접근을 확장시킵니다.
새로운 QIAGEN OmicSoft Lands API는 QIAGEN OmicSoft Lands의 데이터 전체에서 강력한 검색을 가능하게 합니다. 자체적으로 구축한 SQL 데이터베이스에 데이터를 수집할 필요없이 Land 데이터베이스 전체의 모든 메타데이터에서 데이터를 찾을 수 있습니다.
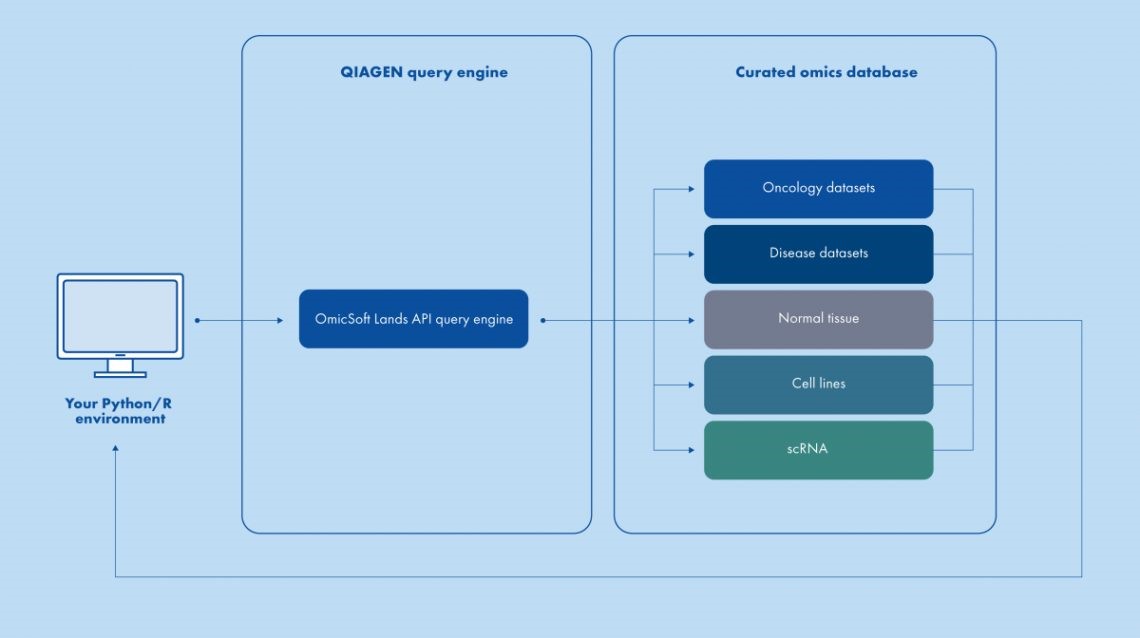
Figure 1. QIAGEN OmicSoft Lands API의 모식도. Python 또는 R 패키지를 통해 SQL 구문을 사용하여 QIAGEN의 선별된 Land 데이터베이스 내에서 일치하는 데이터를 찾습니다. 찾은 데이터는 다운로드 하거나 Python/R에 직접 로드할 수 있습니다.
데이터
QIAGEN OmicSoft Lands API는 여러 프로젝트에서 검색이 가능하도록 메타데이터에 사용되는 단어를 선정하고 관리하여 심도있게 선별된 데이터세트에 직접 접근할 수 있도록 합니다.
데이터 구조
OmicSoft의 구조화된 메타데이터 및 데이터 테이블은 하나 이상의 행으로 검색하여 바이오마커 관련 질문에 대한 답변에 필요한 데이터를 정확하게 찾을 수 있습니다.
QIAGEN OmicSoft OncoLand 및 DiseaseLand 데이터세트는 샘플 수준, 비교 수준, 프로젝트 수준으로 연결시켜서 구조화 및 선별 과정을 거친 메타데이터를 가지고 있고, 이를 아래와 같은 각 샘플의 다양한 측정값들과 연결시켰습니다.
- RNA-seq 유전자 발현(gene, transcript, exon, junction, fusion and mutation)
- 마이크로어레이 발현(probe 및 gene)
- miRNA-seq
- DNA-seq mutation calls
- copy number
- 그 외 다양한 측정값
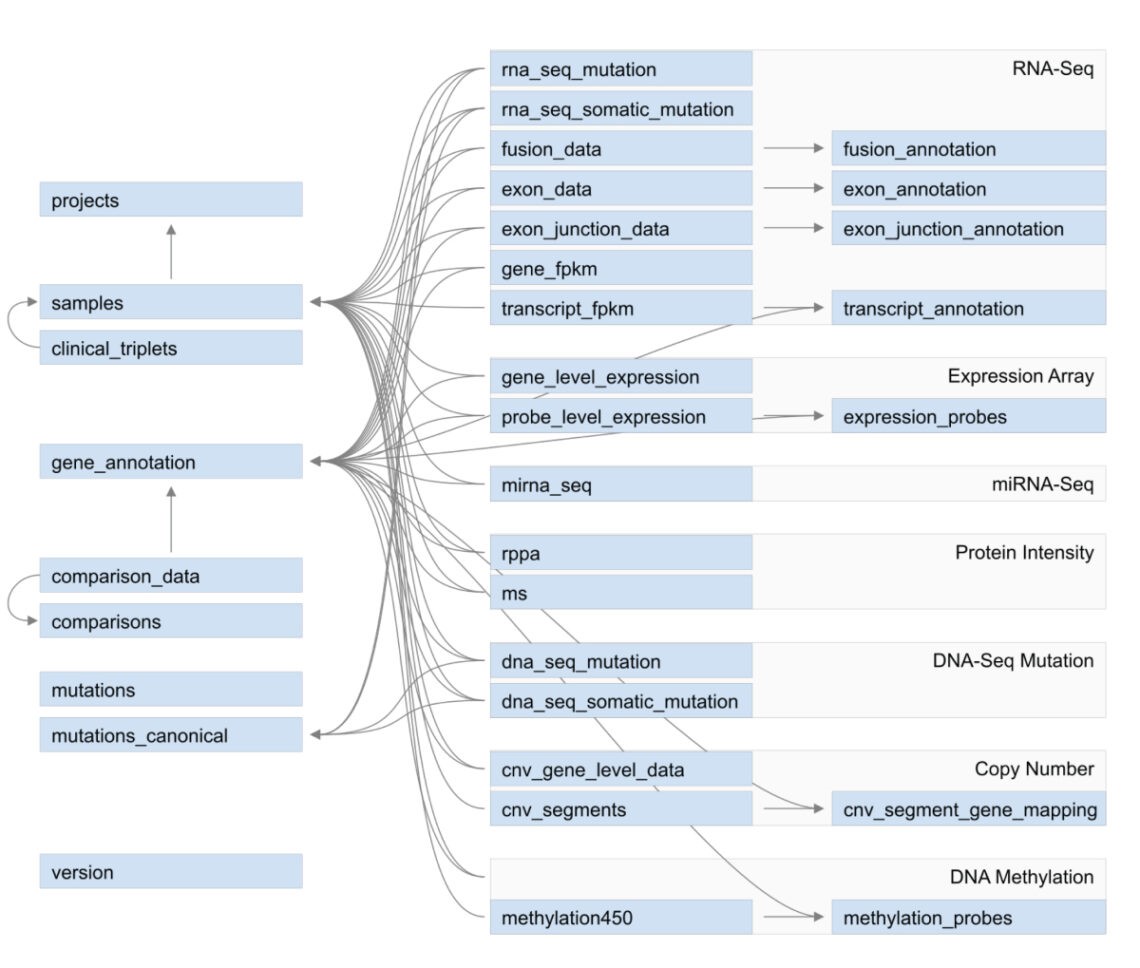
Figure 2. QIAGEN OmicSoft OncoLand 및 DiseaseLand 데이터세트 구조. 선별된 메타데이터(왼쪽)는 프로젝트 및 샘플 수준에서 샘플 그룹 간의 비교가 있는 여러 테이블 내에 표시됩니다. 선별된 각 데이터 유형(가운데)은 연결된 메타데이터, 데이터 값 또는 연결된 주석(오른쪽)에서 필터링할 수 있는 별도의 테이블로 표시됩니다.
QIAGEN OmicSoft Single Cell Land 데이터베이스 내의 단일 세포 데이터세트에는 프로젝트, 샘플, 클러스터, 세포 및 비교 수준에서 메타데이터(유전자 발현과 관련된 각 세포, 차원 감소 분석 등)가 구조화되고 선별되었습니다.
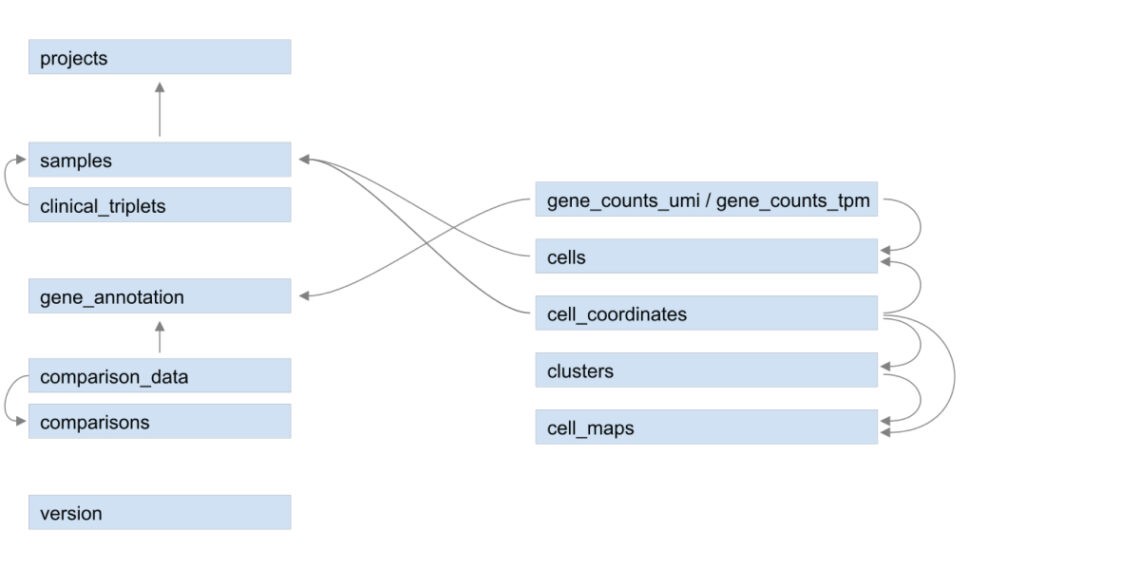
Figure 3. QIAGEN OmicSoft Single Cell Land 데이터베이스의 단일 세포 RNA-seq 데이터세트의 구조. 프로젝트, 샘플 및 비교 수준(왼쪽)에서 선별된 정보 외에도 단일 세포 데이터세트에는 메타데이터와 세포 및 세포 클러스터(오른쪽)를 설명하는 데이터가 포함됩니다.
선별된 데이터에 접근
초기 릴리즈는 아래와 같은 내용을 담고 있으며, QIAGEN OmicSoft DiseaseLand, OncoLand 및 Single Cell Land 데이터베이스 내에서 구독한 데이터세트에 대한 접근을 제공합니다.
- 650,000개 이상의 수동 선별 샘플
- 9,000개 이상의 오믹스 프로젝트에서 대표되는 1,400개 이상의 질병 및 700개 이상의 조직
- 강력한 검색 기능으로 6,000개 이상의 풍부하게 설명된 메타데이터에서 연구 전반에 관련된 샘플의 획득
- 124,000개 이상의 맞춤형 통계 모델 비교를 통해 연구에서 차등 발현 유전자 특징 확인
API 클라이언트 시스템 요구 사항
- Python(버전 3.7 이상) 및 R(버전 3.6.0 이상)을 지원