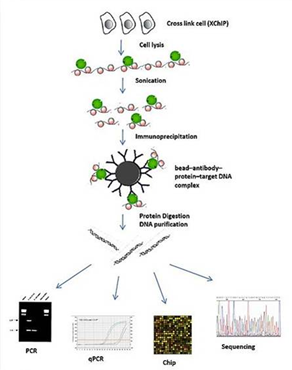
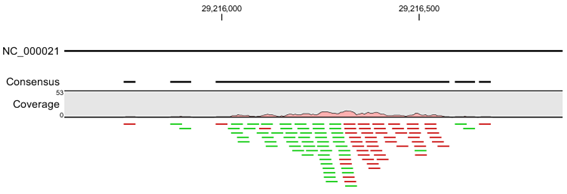
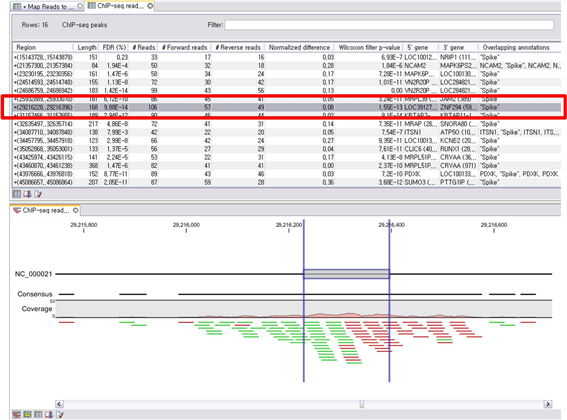
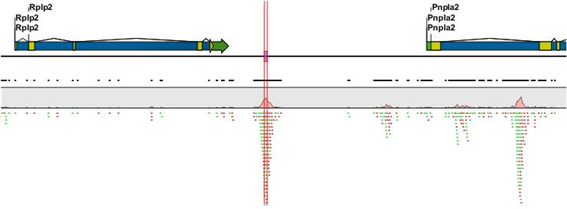
개인 맞춤 의학시대를 가능케 한 NGS(Next Generation Sequencing) 기술로 인해 이제는 더 이상 유전자 서열정보만을 밝히는 것이 큰 의미를 내포하지 않는다. 생명과학 분야의 궁극적인 목표인 생명현상의 이해를 위해서는 쏟아지는 서열정보를 잘 꿰어 그들의 매우 정교한 세포내 역할을 규명해야 한다. (주)인실리코젠에서는 이러한 연구를 위해 필수적으로 요구되는 몇 가지 데이터베이스를 소개하고자 한다.
최근 nature에 발표된 Ancient human genome project에 이용된
전사 조절인자 데이터베이스로 유명한
TRANSFAC을 서비스하고 있는
Biobase는 전문가 리뷰에 의한 생물학적 데이터베이스와 소프트웨어 및 생명과학분야의 분석 서비스에 뛰어난 세계적 선두 기업이다.
1986년 시작되어 1997년 German Research Center for Biotechnology에서 파생되어 설립된 이후로 전사조절인자를 비롯한 유전자 조절 메카니즘 데이터베이스 분야에서 독보적인 위치를 차지해 오고 있다. 의학을 비롯한 제약회사 및 연구기관을 포함한 전세계 수많은 고객에게 서비스를 제공하고 있으며, 생명과학 분야의 다양한 논문에서 현재의 데이터가 인용되고 있다.
Biobase 제품군의 가장 큰 특징은 생물학 전문가들에 의한 데이터의 검토와 수정을 통해 지속적으로 업데이트된다는 것이다. 날마다 논문을 통해 쏟아지는 생명과학 분야의 다양한 데이터를 전문가의 리뷰를 통해
BIOBASE Knowledge Libray(BKL)로 재탄생 시켜 제공하고 있고 이들 데이터의 이해를 극대화 시킬수 있는 ExPlainTM을 서비스 함으로써 drug 혹은 biomarker 개발에 많은 연구자들이 효율적으로 활용 할 수 있도록 하고 있다. 그 서비스 목록은 크게 세 가지로 분류 된다.
1) BKL TRANSFAC
2) BKL PROTEOME
3) HGMD professional
첫 번째,
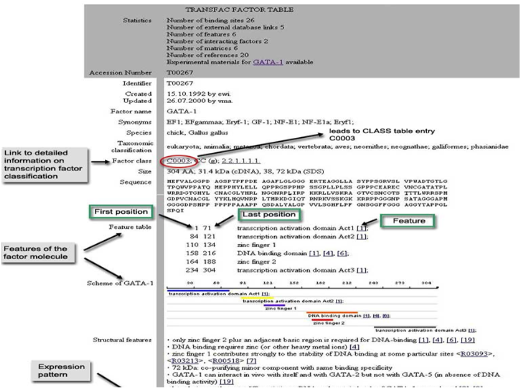
TRANSFAC은 유전자 조절분야에서 세계 유일의 데이터베이스이며 표준이 될 정도의 고품질 데이터를 보장하고 있다. 이러한 평가는 The U.S. Bioinformatics Market의 보고에서도
TRANSFAC®을 주요 생물정보 툴 중 하나로 꼽는 등 세계적으로 높은 평가를 받고 있다. TRANSFAC suite에는 전사 조절인자와 관련된 모든 정보를 담고 있다.
Transcription factor, transcription factor binding site, 그리고 composite elements의 총체적인 정보로 구성되어 있으며, 유전자 돌연변이와 유전자 돌연변이에 관련된 질병에 관한 데이터베이스인 PathoDBTM 그리고 regulatory chromatin domain 정보를 담고 있는 S/MARtDBTM도 포함하고 있다.
두 번째,
PROTEOME은 단백질 수준의 조절, 즉 pathway정보를 제공하고 있다. 6개의 데이터베이스로
YPD(s.cerevisiae), HumanPSD, GPCR-PD, WormPD, MycoPath PD 그리고
PombePD(s.pombe)로 구성되어 기능이 밝혀진 최대한의 단백질을 활용하여 세포내에서의 pathway 조절 메카니즘을 총체적으로 이해 할 수 있도록 정보를 제공하고 있다. 이들 데이터는 관련 질병정보를 비롯한 참조논문과 데이터의 품질 정보를 모두 제공함으로써 다양한 생명과학 분야에서 인용되고 있다.
마지막
HGMD는 human의 유전자 돌연변이 데이터베이스로 유전에 의한
질병관련 정보를 서비스하고 있다. Germ-line 돌연변이 데이터를 중심으로 주어진 유전자와 관련된 돌연변이 정보를 제공하고 있다. 2006년 이후 꾸준한 데이터베이스의 축척으로 2009년 3월
95,000건에 달하는 돌연변이 정보를 보유하고 있으며, 병변을 비롯한 서열정보, 유전체에서의 위치정보, 본래 특성 정보등 상세한 관련 정보를 제공 하고 있다.
앞서 밝힌 내용과 같이 Biobase 제품군은 세포내 발현 조절과 관련된 총체적인 데이터베이스를 제공한다.
전사 수준의 발현조절인 promoter 분석(TRANSFAC), 단백질 수준의 pathway 분석(PROTEOM), 이후 phenotype과 관련된 유전적 질병 정보(HGMD) 등을
제공하며 다양한 생명과학 분야에 고품질의 데이터를 제공하고 있다.
다음 주부터 앞으로 3주 동안, 오늘 간략하게 말씀드린 Biobase 제품군의 세 가지 데이터베이스에 대하여 한 주에 하나씩 좀 더 자세한 내용으로 소개해드릴 예정입니다.
여러분들의 많은 관심 부탁드립니다.
감사합니다.
Posted by 人Co