약물 작용과 효과의 profiling을 위한 ChemEffect Database
- Posted at 2010/06/28 17:39
- Filed under 제품소식
연재 순서
1. PathwayStudio 소개
2. 문헌정보를 활용한 유전자 네트워크 분석
3. Chip 실험 데이터에서의 유전자 네트워크 분석
4. Drug 발굴을 위한 지식 데이터베이스 ChemEffect
약물 작용과 효과의 profiling을 위한 ChemEffect Database
현재 지구상에는 약 800만 종의 화학물질이 존재하고 그 중에서 8만여 종이 상업적으로 생산, 판매되고 있다고 한다. 우리나라에서 사용되는 화학물질의 종류는 약 1만여종으로 매년 증가하고 있다. 이러한 화학물질 가운데 사람의 유전자에 영향을 미치는지 파악하기 위해서는 DNA chip실험을 통한 분석 및 다른 다양한 실험을 통해서 판별이 가능하다.
약제로 개발되는 화학물질의 경우 인체에 부작용을 일으키는지 여부는 실험을 거치지 않고서는 판별할 수 없다. 최근 들어, 독성유전학이라는 분야가 새로운 연구로 각광을 받고 있으며, 산업 현장 및 일상생활속에서 접하고 있는 중금속이 인체의 유전자에 미치는 영향을 파악하고자 하고 있다.
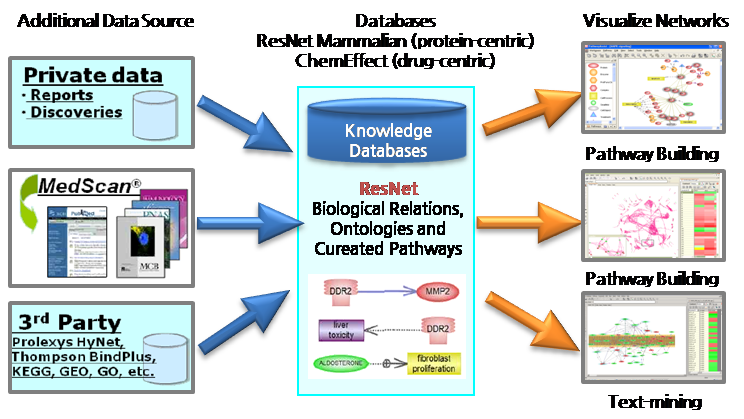
AriadneGenomics사 에서는 이러한 화학물질의 특성 및 유전자에 영향을 미치는 지 여부 등을 기존의 논문 정보와 실험 정보등을 통해서 “ChemEffect” 라는 데이터베이스를 구축하여 서비스를 제공하고 있다.
ChemEffect 데이터베이스는 화학물질인 small molecular가 gene과의 연관관계 및 cellular processes에서의 역할들에 대한 다양한 정보를 담고 있다. 이 정보는 약물의 화학물질에 대해서 독성과 compound 타입에 의한 부작용을 프로파일링 하거나 효소정보를 찾을 때, 독성과 약물 메커니즘 사이의 관계를 결정 지을 때 그리고 대체 물질을 찾을 때 많은 도움이 된다.
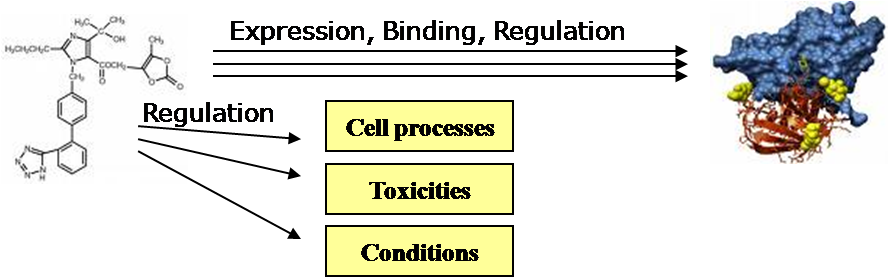
ChemEffect
데이터베이스를 이용하면 아래와 같은 문제들을 빠르게 해결할 수 있다.
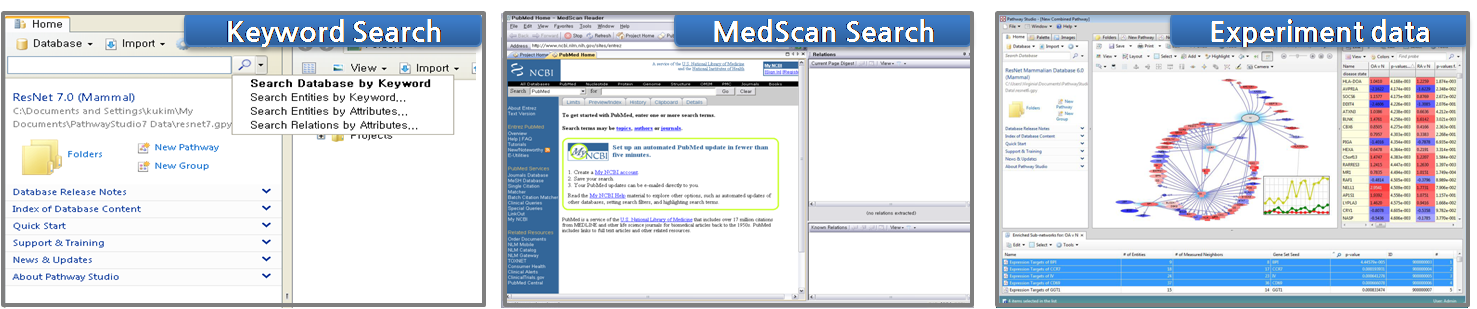
ChemEffect는 NLP(Natural Language Processing) 기술을 응용하여 과학문헌 정보에서생물학적인 정보를 추출하는 MedScan과 추출된 정보를 이용하여 다양한 정보들 간의 네트워크를 그래픽적으로 표현할 수 있는 PathwayStudio로 구성되어 있다.
- 후보 약물과 관련된 pathway와 연결되어있는 질병을 확인할 때,
- Target pathway 또는 Target protein에 영향을 미치는 compound를 발견하고자 할 때,
- Compound에 의해 영향을 받는 Target protein을 찾을 때,
- Compound와 관련 있는 부작용에 대한 연구를 할 때,
- 약효, 독성, drug-drug 작용과 같은 잠재적인 결과와 compound 사이의 관계를 설계 할 때,
ChemEffect 데이터베이스의 구성
ChemEffect는 NLP(Natural Language Processing) 기술을 응용하여 과학문헌 정보에서생물학적인 정보를 추출하는 MedScan과 추출된 정보를 이용하여 다양한 정보들 간의 네트워크를 그래픽적으로 표현할 수 있는 PathwayStudio로 구성되어 있다.
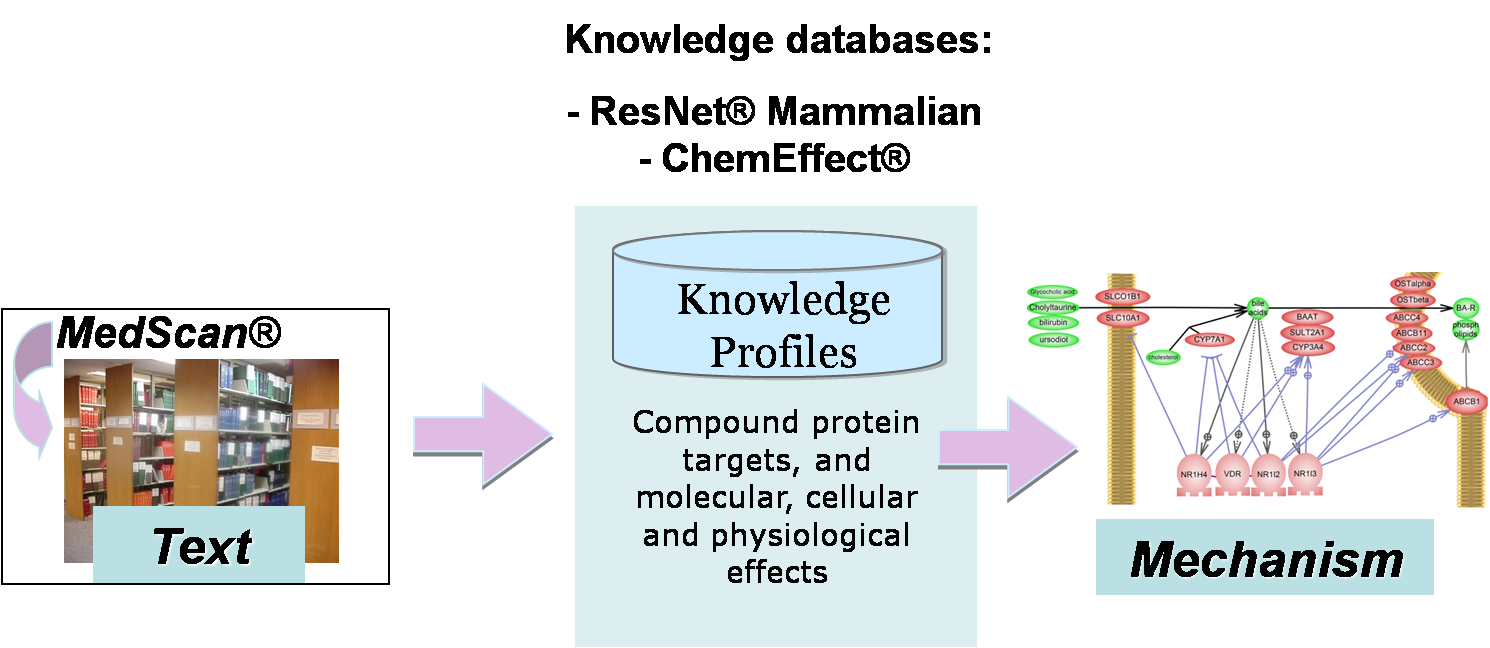
ChemEffect 데이터의 구성
ChemEffect의 데이터는 앞서 말한 것과 같이 MedScan의 텍스트-마이닝 기법을 이용하여 추출된다. Pathway Studio의 기본 데이터베이스인 ResNet Mammalian 데이터베이스와 결합된 ChemEffect에는 NCBI PubMed에 투고된 논문 정보로부터 추출된 Entity, Relations 정보 이외에도 기존에 알려져 있는 Pathway, Ontologies, Annotation 정보들도 함께 포함하고 있어 보다 다양한 정보를 얻을 수 있다.

ChemEffect 데이터베이스를 활용한 Workflow
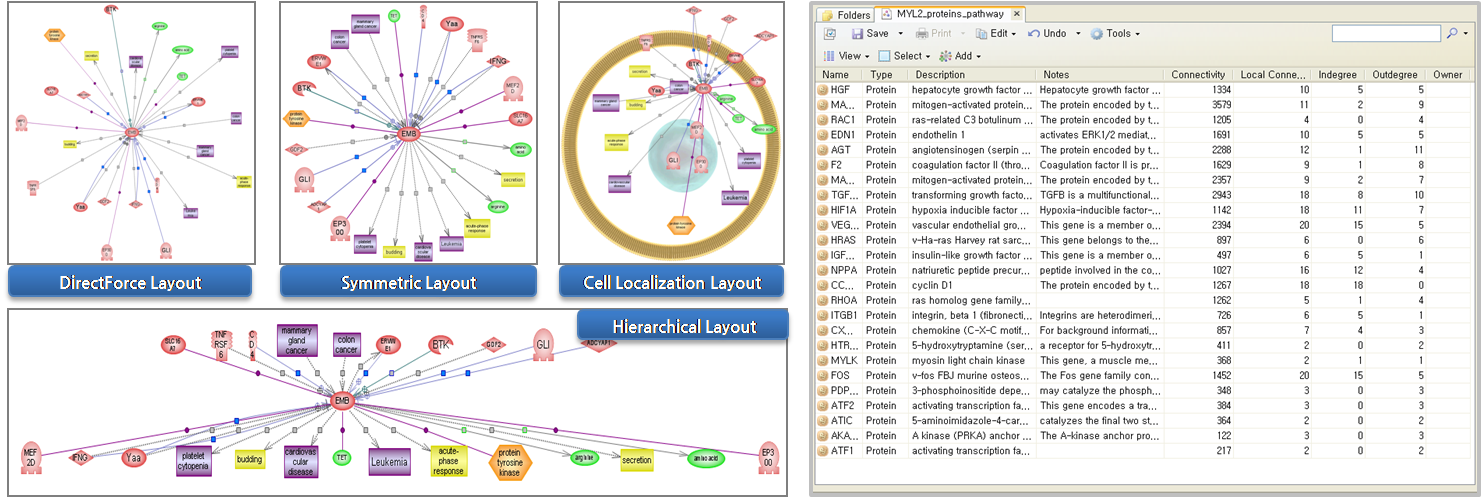
ChemEffect 데이터베이스를 활용해서 1차적으로 보고자 하는 drug 또는 small molecule에 대해 지식 기반의 프로파일링을 수행 할 수 있다. 예를 들어 Sorafenib라는 Small molecule의 1차 검색을 통해서 이 small molecule과 관련 있는(Metabolized by, Directly Inhibits, DownRegulates, UpRegulates) 단백질 정보와 Sorafenib에 의해 Inhibit되고 Activate하는 cell processes에는 어떤 것들이 있는지 프로파일링 정보를 얻을 수 있다. 모든 정보에 대해서는 이를 뒷받침하는 문헌 정보와 링크가 되어 있어 바로 확인이 가능하므로 분석된 데이터에 대한 신뢰성이 상당히 높다고 할 수 있다.

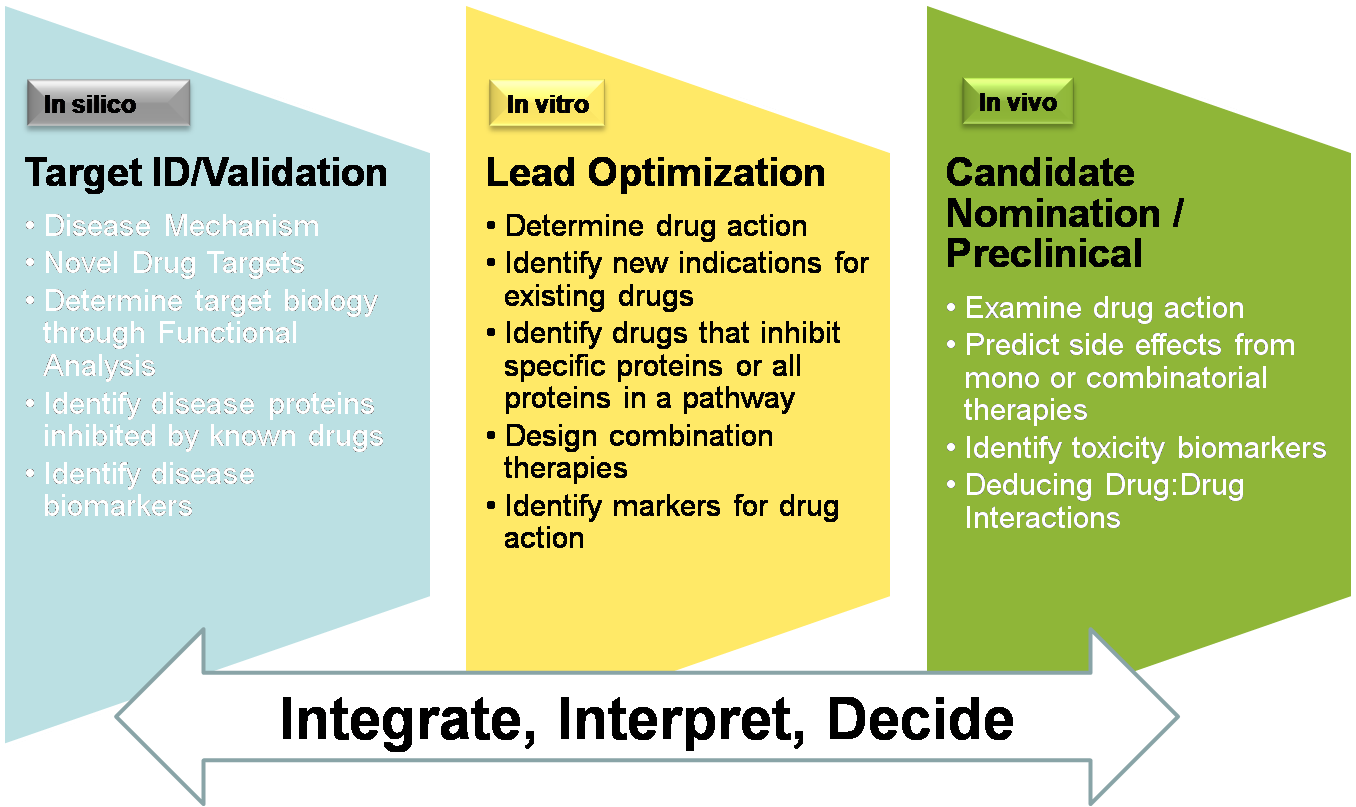
더 나아가서는 Drug Discovery를 할 때 다양한 Application에서 ChemEffect 데이터베이스를 사용할 수 있다. In silico 단계에서 Target을 validation 할 때, In vitro 단계의 Lead Optimization, In vivo 단계에서의 Candidate Nomination/Preclinical 이 세 가지 모든 과정을 통합하고 해석하고 마지막으로 최종 결정에 이르기까지 유용하게 응용 될 수 있다.
Posted by 人Co
- Tag
- ChemEffect, Chip, Database, Drug, Drug discovery, Insilico, insilicogen, MedScan, NLP, PathwayStudio, profiling, Sorafenib, 데이터베이스, 독성, 문헌정보, 약물 메커니즘, 약물작용, 인실리코젠
- Response
- No Trackback , No Comment
- RSS :
- https://post-blog.insilicogen.com/blog/rss/response/77